第280回「生体分子を設計するAI 診断・創薬 大きく加速」
従来のAI(人工知能)は特定のタスクごとにデータを学習させるタスク特化型だったが、チャットGPTに代表される生成AIは、さまざまな問いに対して回答を生成でき、高い汎用性を示す。そこでは、大規模で多様なデータで事前学習し、その後の追加学習などにより多様なタスクに適応できる「基盤モデル」が用いられている。基盤モデルのうち、人間の言語を学習したものは大規模言語モデル(LLM)と呼ばれる。
配列を学習
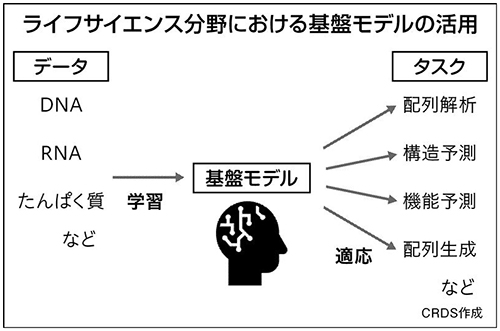
近年、ライフサイエンスの分野では、人間の言語の代わりにデオキシリボ核酸(DNA)やリボ核酸(RNA)、たんぱく質といった生体分子の配列を学習した基盤モデルが登場している。これらの基盤モデルは生体分子の配列解析、構造や機能の予測、新たな生体分子の設計などの多様なタスクを実行できる。
中でも、「タンパク質言語モデル」は急速に発展してきた。2024年に米エボリューショナリースケールから発表されたESM3は、27億8,000万個のたんぱく質のデータで学習させたタンパク質言語モデルである。たんぱく質の配列、構造、機能を高い精度で予測でき、自然界には存在しない新たな蛍光たんぱく質の設計にも成功した。
同年、米アーク研究所と米スタンフォード大学のチームから発表されたEvoは、ゲノム(ある生物が持つDNAの配列全体)の基盤モデルとして注目される。Evoは270万個の細菌などのゲノムを学習しており、たんぱく質の機能予測をはじめとした幅広いタスクに成功しただけでなく、細菌のゲノムを最終的には丸ごと生成できる可能性も示された。さらに25年2月、ヒト、植物、酵母などのゲノムも学習したEvo2が発表され、ヒトの遺伝子変異の病原性の予測にも成功した。
応用多岐に
これらの基盤モデルは、ライフサイエンス研究に新たなアプローチを提供するとともに、疾患の診断や新薬の設計、農水畜産物の改良など、その多岐にわたる応用を大きく加速させる。大手製薬企業や巨大IT企業による大規模な投資を背景に、米欧中のスタートアップや大学を中心に研究開発が精力的に進められている。
一方で、予測、設計した新たな分子や細胞などが、人々の健康や地球環境へ与える影響に関するルール作りの議論も必要になるであろう。基盤モデルは、これからのライフサイエンス分野の中核を担い得る技術であり、わが国の研究力強化の観点から優先度の高いテーマである。
※本記事は 日刊工業新聞2025年3月14日号に掲載されたものです。
<執筆者>
戸田 智美 CRDSフェロー(ライフサイエンス・臨床医学ユニット)
東京大学大学院農学生命科学研究科修士課程修了。ライフサイエンス関連のテーマを対象に調査・提言を実施。修士(農学)。
<日刊工業新聞 電子版>
科学技術の潮流(280)生体分子を設計するAI、診断・創薬を大きく加速(外部リンク)
