
研究総括 胡桃坂 仁志
(東京大学 定量生命科学研究所 教授)
研究期間:2019年10月~2025年3月
グラント番号:JPMJER1901
ヒトを含めた真核生物において、ゲノムDNAはたんぱく質が結合した「クロマチン」と呼ばれる分子複合体として折りたたまれて細胞核内に収納され、ゲノムDNAの転写、複製、修復、組み換えといった機能は、クロマチンの適切な折りたたみ構造によって制御されています。クロマチン構造の破綻は様々な疾病と関連する可能性が指摘されており、この機能の解明は生命の遺伝情報利用の根幹に迫るとともに、関連疾患においてこれまでにない新たな治療法の確立に大きく貢献することが期待されます。
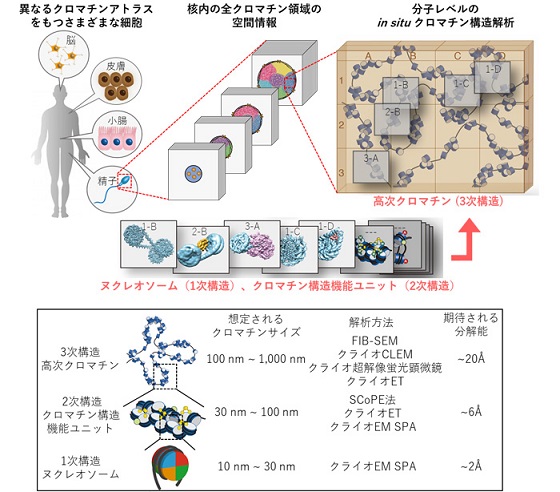
本プロジェクトでは、遺伝情報の本体であるゲノムDNAがどのように細胞核内に収納されているのか、その構造と機能を解き明かすことで、真核生物がDNAを利用する仕組みについて新たな概念を創出することを目指します。具体的にはクロマチンの折りたたみ構造地図を「クロマチンアトラス」と定義し、近年著しい技術革新を遂げているクライオ電子顕微鏡技術を中心に据えて、その構造と機能の相関性を網羅的に解明することを目指します。
研究グループ
・クロマチン構造研究グループ
・有機合成化学研究開発グループ
・表現型解析グループ
研究成果の概要
クロマチンは、DNAがヒストン複合体に巻き取られたヌクレオソーム(1次構造)を基本単位とした構造体である。近年では、このヌクレオソーム自体にも多様な立体構造があることが明らかになっている。この1次構造は多くのヌクレオソーム結合因子群を伴って集積し、クロマチン構造機能ユニット(2次構造)構築し、さらに2次構造間での相互作用が巨大な高次クロマチン(3次構造)を形成する。こうした多様なクロマチン構造により、遺伝情報の高度な制御が可能になっていると考えられている。
本プロジェクトでは、新たなクロマチンアトラスの構造情報を多数提供するとともに、クロマチンアトラス可視化のための新技術の開発に成功した。これにより、多様なDNA機能発現「場」におけるクロマチンの役割が明らかになるとともに、生体内クロマチンアトラスの構築に向けた重要な一歩を踏み出した。
研究成果
クロマチンアトラスの機能領域を構成するクロマチン基盤構造の解明
クロマチン構造は、多様な核内イベントの制御に深く関与している。本プロジェクトでは、細胞核内の様々なDNA機能発現「場」におけるクロマチン構造機能ユニットの基盤構造に着目し、再構成技術により調製されたクロマチンを、クライオEM単粒子解析による高分解能構造解析を行った。
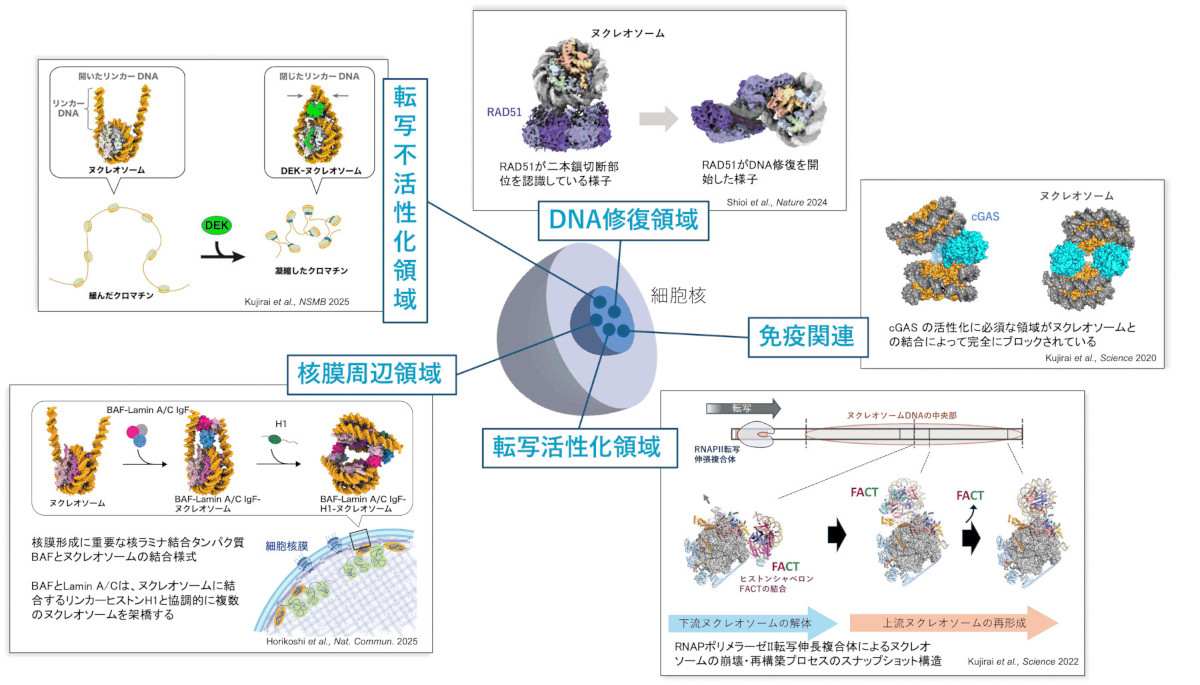
まず、自然免疫におけるDNAセンサー cGAS の不活化機構について、cGAS とヌクレオソームの複合体の立体構造解析を通じて解明した(Kujirai et al., Science 2020)。本成果は、自然免疫の中心的問である自己と非自己のDNAを区別する仕組みについての重要な知見を与えたのみならず、cGASが生体内クロマチン構造を形成する様子が初めて明らかにすることができた。cGASとヌクレオソームによって形成されるサンドイッチ型の構造は、クロマチンアトラスの重要な基盤構造の1つとなりうると考えられる。本発見は、cGASが関連する広範な疾患の原因解明や治療法確立のために重要な情報を提供すると考えられる。
また、RNAポリメラーゼ II(RNAPII)と転写伸長因子が結合した転写伸長超分子複合体とヒストンシャペロン FACTの協調によるクロマチン転写の分子機構を解析することに成功した(Kujirai et al., Science 2022)。ヌクレオソームと転写伸長複合体はいずれもクロマチンアトラスにおいて主要なクロマチン構造体であり、クロマチンアトラスとゲノム機能の相関を理解する上で、重要なターゲットである。本成果はクロマチンアトラスの1次構造に加え、2次構造である機能的ユニットの構造を明らかにするものとなった。
さらに、相同組換えの主要因子である RAD51 とヌクレオソームの複合体の構造群を決定し、クロマチン上で DNA 損傷がどのように認識され、修復が開始されるのかという長年の疑問であった重要な課題に対する新たな知見を提供した(Shioi et al., Nature 2024)。これらの発見は、クロマチン上におけるRAD51を中心とした相同組換えの開始ステップを分子レベルで明らかにした。
これらの成果は、真核生物のクロマチンでのDNA修復の理解を深めるのみならず、がん研究に対しても新たな視点を提供する。がん患者ではRAD51のN末端領域(NLD)の変異が多く見つかっていることから、本研究における構造的および機能的な特性の解明は、がん細胞におけるRAD51のNLDを標的とする新規治療法開発への道を開くことが期待される。その他にも、がん関連タンパク質 DEK とヌクレオソームの複合体構造(Kujirai et al., Nat Struct Mol Biol 2025)や細胞核膜近傍に局在する核ラミナ結合タンパク質 BAF とヌクレオソームの複合体構造(Horikoshi et al., Nat Commun 2025)を含む多数のクロマチン複合体構造を明らかにした。
本プロジェクトにより、新規のクロマチン構造ユニットを含むクロマチンの立体構造ライブラリーを作成することに成功した。このライブラリーは、核内のゲノム機能を理解する上で不可欠な基盤的マテリアルとなり、今後のクロマチン研究の発展に大きく貢献することが期待される。
- Horikoshi N., Miyake R., Sogawa-Fujiwara C., Ogasawara M., Takizawa Y., Kurumizaka H. Cryo-EM structures of the BAF-Lamin A/C complex bound to nucleosomes. Nat Commun. 16, 1495 (2025) doi: 10.1038/s41467-025-56823-9.
- Kujirai T., Echigoya K., Kishi Y., Saeki M., Ito T., Kato J., Negishi L., Kimura H., Masumoto H., Takizawa Y., Gotoh Y., Kurumizaka H. Structural basis of nucleosome binding by DEK that facilitates H3K27 trimethylation in chromatin. Nat Struct & Mol Biol. (2025) doi: 10.1038/s41594-025-01493-w.
- Shioi T., Hatazawa S., Oya E., Hosoya N., Kobayashi W., Ogasawara M., Kobayashi T., Takizawa Y., Kurumizaka H. Cryo-EM structures of RAD51 assembled on nucleosomes containing a DSB site. Nature, 628, 212-220 (2024) doi: 10.1038/s41586-024-07196-4.
- Ehara H*., Kujirai T*., Shirouzu, M., Kurumizaka, H., & Sekine, S.I. Structural basis of nucleosome disassembly and reassembly by RNAPII elongation complex with FACT. Science, 377, 6611 (2022) doi: 10.1126/science.abp9466.
- Kujirai T*., Zierhut C*., Takizawa Y., Kim R., Negishi L., Uruma N., Hirai S., Funabiki H., Kurumizaka, H. Structural basis for the inhibition of cGAS by nucleosomes. Science, 370, 455-458 (2020) doi: 10.1126/science.abd0237.
細胞核から抽出したクロマチンアトラスの機能的クロマチン構造ユニットの同定と構造解析
ヒト細胞核から直接取り出したクロマチン構造機能ユニットおよび高次クロマチン構造の解析を行った。
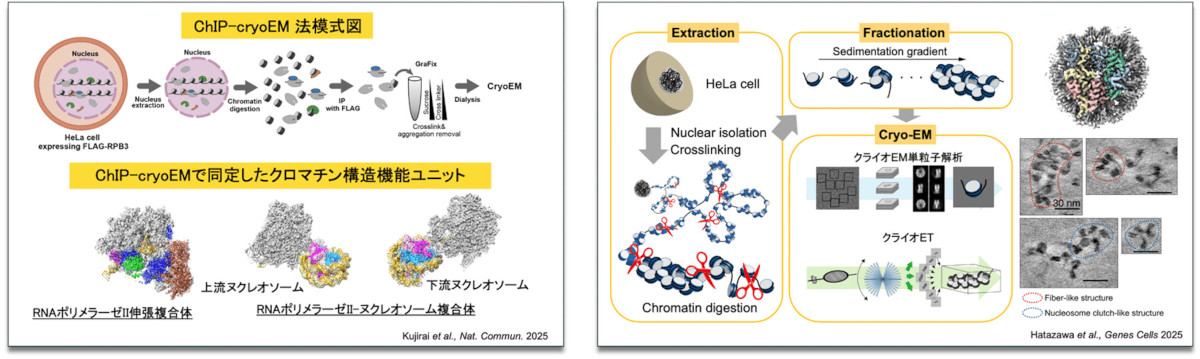
核内におけるクロマチンの構造動態をより深く理解するために、クロマチン結合タンパク質複合体をヒト細胞から免疫沈降法を用いて単離し、クライオEMによって立体構造を同定する革新的な新技術「ChIP-CryoEM法」を確立した。このアプローチにより、ヒト細胞内でゲノムDNAを転写中の RNAPII 伸長複合体や、さまざまな転写因子やヒストン結合タンパク質が結合する巨大な機能的クロマチン複合体の抽出に成功し、クライオEMによる構造解析を実現した(Kujirai et al., Nat Commun 2025)。
また、高次クロマチン構造を可視化するため、HeLa細胞よりクロマチンを抽出することにより、クライオEM構造解析を行った。クロマチンを自然に近い状態で抽出し、基本単位のヌクレオソームからクロマチン構造機能ユニットまで幅広く分けた。このように得られたクロマチン構造機能ユニットであるポリヌクレオソームのクライオET解析を行った結果、ファイバー様のクロマチン構造やヌクレオソームが集合したようなクロマチンの構造を可視化することに成功した(Hatazawa et al., Genes Cells 2025)。
本プロジェクトで開発した新規のクライオEM関連技術は、従来試験管内再構成に頼っていた構造生物学の概念を覆す革新的な技術であり、クロマチン分野だけではなく構造生物学研究で幅広く使われる標準的な研究手法へと発展することが期待される。
- Hatazawa S., Fukuda Y., Kobayashi Y., Negishi L., Kikkawa M., Takizawa Y., Kurumizaka H. Cryo-EM structures of native chromatin units from human cells. Genes Cells., 30, e70019 (2025) doi: 10.1111/gtc.70019.
- Kujirai T., Kato J., Yamamoto K., Hirai S., Fujii T., Maehara K., Harada A., Negishi L., Ogasawara M., Yamaguchi Y., Ohkawa Y., Takizawa Y., Kurumizaka H. Multiple structures of RNA polymerase II isolated from human nuclei by ChIP-CryoEM analysis. Nat Commun., 16, 4724 (2025) doi: 10.1038/s41467-025-59580-x.
クロマチンアトラス可視化のためのクライオ蛍光顕微鏡開発
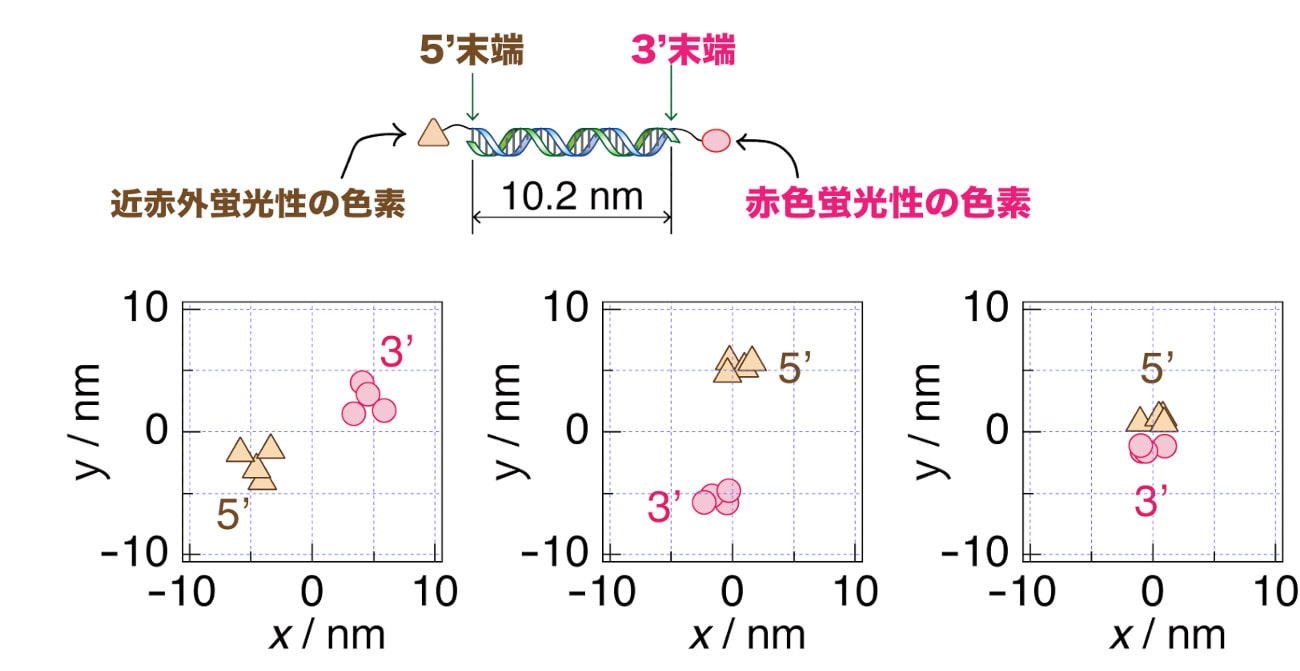
細胞核内クロマチンアトラスの基盤構造の空間情報解析を可能にする高分解能クライオ蛍光顕微鏡を開発した。この技術の確立に向け、まず世界で初めてヌクレオソーム同士の距離を識別できる分解能5 nmを達成した(Furubayashi et al., J Phys Chem Lett 2020)。さらに、観察の最適化に向けた技術改良を重ね(Ishida et al., Opt Lett 2021;Kamiya et al., Rev, Sci. Instrum. 2022;Otomo et al., J. Phys. Chem. B 2023)、細胞の凍結切片のクライオ蛍光顕微鏡画像の取得に成功した。
今後は、細胞核内の特定のクロマチン高次構造情報を得るために、クライオ蛍光顕微鏡とクライオ電子顕微鏡を組み合わせた解析を進め、細胞核内のクロマチン状態を詳細に解明する。この先進的な技術は、クロマチンアトラスの高次構造解析だけでなく多様な標的への応用が期待される。
- Otomo K., Dewa T., Matsushita M., Fujiyoshi S. Cryogenic single-molecule fluorescence detection of the mid-infrared response of an intrinsic pigment in a light-harvesting complex. J. Phys. Chem. B, 127, 4959-4965 (2023) doi: 10.1021/acs.jpcb.3c00284.
- Kamiya N., Kuramoto K., Takishima K., Yumoto T., Oda H., Shimi T., Kimura H., Matsushita M., Fujiyoshi S. Superfluid helium nanoscope insert with millimeter working range. Rev, Sci. Instrum., 93, 103703 (2022) doi: 10.1063/5.0107395.
- Ishida K., Natuse K., Mizouchi Y., Ogawa Y., Matsushita M., Shimi T., Kimura H., Fujiyoshi S. Variable immersion microscopy with a high numerical aperture. Opt. Lett., 46, 856-859 (2021) doi: 10.1364/OL.416006.
- Furubayashi T., Ishida K., Kashida K., Nakata E., Morii T., Matsushita M., Fujiyoshi S. Nanometer accuracy in cryogenic far-field localization microscopy of individual molecules. J. Phys. Chem. Lett.,10, 5841-5846 (2020) doi: 10.1021/acs.jpclett.9b02184.
精子細胞におけるクロマチンアトラスの解明
本プロジェクトでは、クロマチン構造が大きく変化する精子形成の各段階におけるクロマチンアトラスに注目した。哺乳類の精子は、成熟精子へと分化する過程で90%以上のヒストンが高塩基性タンパク質であるプロタミンに置き換わることで核凝集が起こり、精子核が形成される(Okada et al., Genes Genet Syst. 2022; Okada et al., Curr Opin Cell Biol. 2022) 。本プロジェクトでは、この核凝集過程におけるクロマチンの微細な構造変化を追跡するため、精子形成の各段階における精子細胞を高純度に調製し、詳細な観察を行うことに成功した(Fujiwara et al., Cytometry A 2023)。
今後は本プロジェクトで新たに確立した技術を、クライオEM技術およびクライオET技術と融合させることで、未だ多くの謎に包まれている精子形成に伴うクロマチン凝集過程における構造変動と細胞機能の変化のメカニズムを解明していく。
さらに、精子核凝集過程におけるクロマチン凝集の破綻が不妊の要因となることが知られている。例えば、ヒストンの脱落やプロタミンへの置換が不完全であることにより、精子クロマチン内のヒストンとプロタミンの比率が異常になることは、男性不妊において最も頻繁に認められる表現型の一つである。本プロジェクトを通じて得られる精子のクロマチン構造情報は、男性不妊の病態理解に大きく貢献することが期待される。
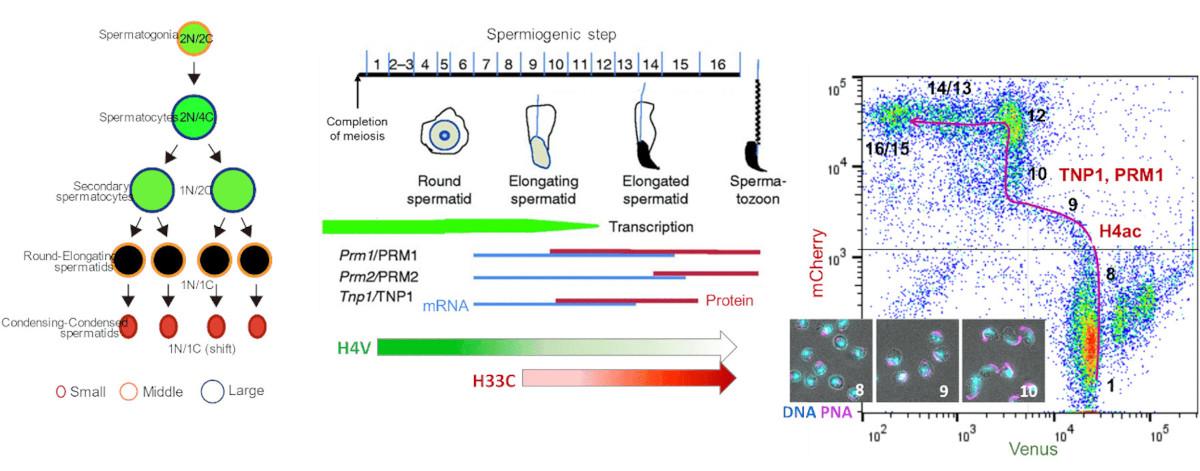
- Fujiwara Y., Hada M., Fukuda Y., Koga C., Inoue E., Okada Y. Isolation of stage-specific spermatogenic cells by dynamic histone incorporation and removal in spermatogenesis. Cytometry Part A. 105, 297-307 (2023) doi: 10.1002/cyto.a.24812.
- Okada Y. Sperm chromatin condensation: epigenetic mechanisms to compact the genome and spatiotemporal regulation from inside and outside the nucleus. Genes Genet Syst. 97, 41-53 (2022) doi: 10.1266/ggs.21-00065.
- Okada Y. Sperm chromatin structure: Insights from in vitro to in situ experiments. Curr Opin Cell Biol. 75, 102075 (2022) doi: 10.1016/j.ceb.2022.102075.