
研究総括 佐藤 匠徳
((株)国際電気通信基礎技術研究所 佐藤匠徳特別研究所 所長)
研究期間:2013年10月~2019年3月
特別重点期間:2019年4月~2020年3月
グラント番号:JPMJER1303
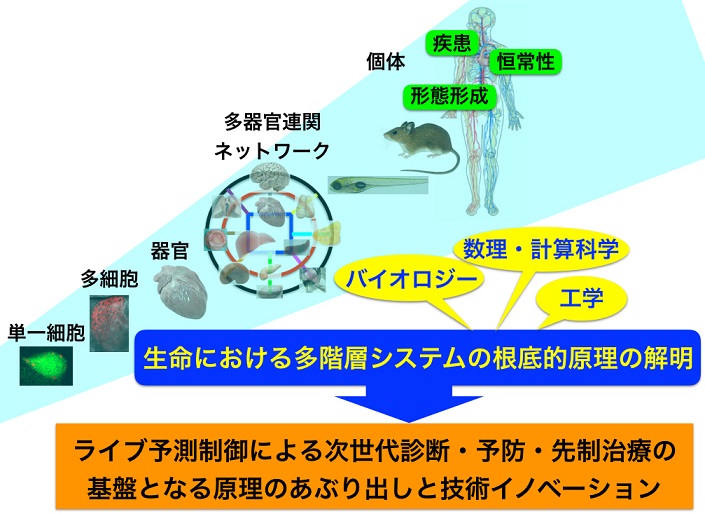
人の体はおよそ37兆個の細胞からなります。個々の細胞は組織を成して器官を形成し、それぞれの器官は「多器官連関ネットワーク」という、器官同士のネットワークを介して協調的にはたらくことで、人の体の恒常性を維持しています。近年、このような多階層システムの構築・維持・破綻のメカニズに関する研究が盛んに行われており、個々の現象に関する詳細な知見の蓄積が進んでいます。しかし、未だに全体像の統合的理解には至っていません。また、生命現象においては各階層の関係が非線形であるため、多階層システムの全体を支配する原理のあぶり出しや、システム全体の理解に基づく個々の現象の説明や予測には限界がありました。
本プロジェクトは、多階層システムの「根底にある支配的メカニズム」を解明し、さらに多階層システムの破綻と疾患との因果関係を明らかにします。そして、多階層システムが統合的に捕らえられる「いきものの設計図」を作成します。さらに、多階層システムにおける時空間の関連性を利用した予測制御に必要不可欠な技術的要素の積み上げからイノベーションを創出します。この目的を達成するために、ゼブラフィッシュやマウスといった動物モデルに加え、人の検体も利用して研究を展開しています。また、各階層が非線形な関係にある多階層システムを扱うために数理・計算科学や工学といった異分野との融合も図りながら研究を推進しています。
本プロジェクトから創出される予測制御技術により、生命活動の状態観測・計測とライブ(実時間)で連動させることが将来的に可能になれば、より効率的で効果的な疾患の診断・予防・先制治療方法の開発につながります。また、生命のようなソフトでゆらぎの多い多階層システムの「設計図」やその予測制御技術は生命科学や医学のみならず、ロボティックスや情報通信といった他の領域におけるイノベーションへもつながる期待されます。
研究成果の概要
研究
これまでのバイオロジーや医学の研究では、体の部分的な研究が主でありました。しかし、本プロジェクトでは、生体の機能や疾患(モデル)を、全身レベルで網羅的に解析することで、個体レベルでの生体機能制御の俯瞰的根本原理をあぶり出し理解することを軸に研究を展開しました。また、これらの基礎研究であぶり出される新たな概念、情報(データ)、ツールを活用した革新的技術の創出をも目指しました。その結果、本研究から以下の成果を得ました:
- A. ゼブラフィッシュのモデルでのライブ予測制御の実現
- B. ゼブラフィッシュの全身レベルでの生体反応の網羅的解析
- C. 個体レベル特有の「死」の制御メカニズム解明
- D. 全身網羅的な多器官連関ネットワークの解明
- E. 全身網羅的な多器官連関ネットワークの制御にかかる新規の器官特異的分泌因子の発見
- F. 全身網羅的な多器官連関ネットワークから発掘されたその他の予防・治療ターゲット
- G. 全身網羅的な多器官トランスクリプトームを活用したバーチャル創薬・バーチャル治験 を可能にするAIシステム(humanized Mouse DataBase: hMDB)の構築
- H. 世の中に存在する全医薬品の全ドラッグリポジショニング標的辞書のAIによる作成
- I. より精度の高い医薬品の適応症の網羅的予測及び作用メカニズムを見える化できるAIシステムの開発
- J. バーチャル全身全細胞トランスクリプトーム地図の作成と活用
これらの成果は、ある時点での生命活動の状態から将来の状態を予測し、将来の状態を自在に制御できる技術のイノベーションにつながります。さらには、これらの予測から制御といった一連の操作を生命活動の状態観測(計測)とライブ(実時間)で連動させることが可能になれば、より効率的で効果的な疾患の診断・予防・先制治療方法の開発につながることも期待されます。我々はこのようなシステムを「Virtual Live Clinics」と呼び、この実現に向けて研究開発を展開しています。
知財化
研究成果の知財化は積極的に行い、本プロジェクトの成果が関わる特許出願は、合計11(内1つは2020年1月中に出願完了予定)になります。その内、5つが日本で登録済みで、現在海外(US, EP, CN)で審査中です。
事業化
本プロジェクトの成果を特許化し、研究総括の佐藤匠徳がファウンダー(兼代表取締役)でKarydo TherapeutiX株式会社を、2015年10月5日に起業しました。現在、様々な業種の大手企業との共同研究開発を展開しています。
完全独立型若手グループリーダーの育成
佐藤ライブ予測制御プロジェクトの主たる研究目的の一貫として、独立した若手2名(河岡慎平、柳谷耕太)をグループリーダーとして採用しました。研究・人材資金は佐藤ライブ予測制御プロジェクトの予算から分配し、予算執行、研究テーマの選定、研究の実施は、佐藤ライブ予測制御プロジェクトから独立して展開してもらうことで、真の独立PIとしての機会を与えました。河岡慎平は「個体レベルでのがん研究」において独立したPIとしての論文をSingle corresponding authorとして3報(Oncotarget, Disease Models&Mechanisms, Nature Communications)に発表しました。佐藤ライブ予測制御プロジェクト終了に伴い、河岡慎平は京都大学ウイルス・再生医学研究所に独立型准教授として着任しました。柳谷耕太は「細胞内オルガネラ量の調節機構」の研究を実施し、佐藤ライブ予測制御プロジェクト終了に伴い、九州大学 生体防御医学研究所 細胞機能制御部門 炎症制御学分野の准教授として着任しました。
アウトリーチ活動
本プロジェクトの研究成果の論文で、主だったものは、ATRおよびJST HPからプレスリリースを出し、各種メジャーメディアにも取り上げて頂きました。また、研究総括の佐藤匠徳が、国内外の招待講演(計32講演 内3は予定)で本プロジェクトの成果を発表しました。さらに、ATRのオープンハウスで一般の方々への講演およびポスター発表も行いました。国際シンポジウムも2つ開催しました。
研究成果
研究
A. ゼブラフィッシュのモデルでのライブ予測制御の実現生体のライブ予測制御のPOCに適した対象候補の一つとして、「生体内の細胞の現在のかたちの計測から、その細胞の将来の運命をライブで予測し、細胞のかたちを人為的に操作することで、細胞の将来の運命を意図的に変える(つまり制御)」ことを選択しました。
1665年、イギリスの自然哲学者のロバート・フックは、当時開発されたばかりの顕微鏡で初めて細胞を観察しました。そして、19世紀には「すべての生き物は1つ、あるいは複数の細胞からなる」という「細胞説」が提唱されました。それ以来、さまざまな細胞が観察され、われわれ人間を含む動物、そして植物は、さまざまな「かたち」をした細胞からなり、細胞のかたちはそれぞれの機能や状態(例えば分裂状態)を決定することが、現代の生物学の教科書には記載されています。そして、近年では、培養中の細胞のかたちを人為的に変える技術により、かたちが機能に与える影響やメカニズムの詳細が明らかになりつつあります。しかし、細胞が過去に持っていたかたちの情報が記憶され、そのかたちを失った後の将来に、その形状記憶が呼び戻され、細胞の機能や運命に影響を及ぼすのかは不明でした。
そこで、もし、そのような現象が存在するのであれば、それを利用し、現在の細胞のかたちから、その細胞の将来の状態を予測すること、また、細胞のかたちを人為的に操作することにより、細胞の将来の運命を操る技術、つまり本プロジェクトの目的の一つである「ライブ予測制御」のPOCを確立することにつながると考えました。
また、この仮説が立証されれば、一細胞レベルでの「形状履歴記憶・記憶呼び出しシステム」という、細胞生物学の分野における、新しい概念の発見につながります。さらに、その根本にある分子メカニズムを解明することで、細胞生物学、および関連分野における、細胞の新たな作動機序の知見が得られると考えました。
そこで、我々は、ゼブラフィッシュの生体内における神経幹細胞であるV2細胞の不均等分裂をモデルに、ライブイメージング、遺伝学、数理モデル、機械学習、計算科学、フェムト秒レーザーによる細胞操作技術、などを駆使して、1)「形状履歴記憶・記憶呼び出しシステム」という新しい概念の確立、2)この概念の分子機構の発見、3)この現象を活用したライブ予測制御のPOC、を達成しました。
成果論文
- T. Akanuma, C. Chen, T. Sato, R.M.H. Merks, T.N. Sato (2016) Memory of cell shape biases stochastic fate decision-making despite mitotic rounding. Nature Communications, 7:11963.
- S. Kozawa, T. Akanuma, T. Sato, Y.D. Sato, K. Ikeda, T.N. Sato (2016) Read-time prediction of cell division timing in developing zebrafish embryo. Scientific Reports, 6:32962.
- T. N. Sato, R. M. H. Merks (2017) Shaping the cell fate. Cell Cycle, 16:149-150.
B. 循環器系の機能不全にともなう低酸素非依存的な生体反応の全身地図作成
ヒトを含む全ての多器官をもつ動物において、個体形成の過程で、心臓・血管系(循環器系)が最初に形成されます。それは、循環器系が他の器官に先立って形成されることで、他の器官へ、酸素や栄養といった、ほぼ全ての器官の形成、および機能獲得・維持に必須のものを運搬する役目を担うからと一般的には考えられています。したがって、個体種の進化の過程で、循環器系は、古くから存在し、保存されてきている、個体形成および維持において非常に重要な器官の一つであることがわかります。そして、その最も重要な機能が、酸素や栄養を他器官へ運搬し、循環させるという機能だと言われています。
しかし、脊椎動物の代表的な実験動物モデルの一つである、ゼブラフィッシュでは、循環器系が形成されなくとも、肝臓やその他多くの器官が、ある程度は形成され、個体形成のある程度の時期までは、機能的にも問題なく働きます。このことから、進化的に、および個体形成という観点からも、循環器系が個体形成で最初に形成される理由は、酸素・栄養の運搬以外にあるのではないかと考えました。
そこで、個体形成で最初に形成される循環器系の、全身機能を支える動物種を超えた根本的なメカニズムを明らかにするために、循環器系の機能不全にともなう低酸素非依存的な生体反応の全身地図の作成に取り組みました。そして、この成果により、全身・個体バイオロジーにおける、概念的発見が得られると考えました。
その結果、全身の各器官で起こる遺伝子発現の変化をカタログ化し、循環器系の役割の全身地図を作成することに成功しました。また、この全身地図から、嗅覚―循環器系、肝臓―循環器系、といったこれまで知られていなかった臓器間連関とその分子メカニズムも明らかになりました。
成果論文
- N. Takada, M. Omae, F. Sagawa, N.C. Chi, S. Endo, S. Kozawa, T.N. Sato (2017) Re-evaluating the functional landscape of the cardiovascular system during development. Biology Open, 6: 1756-1770.
C. 個体レベル特有の「死」の制御メカニズム解明
「死」の制御機構の解明は、生物学・医学の根幹をなす研究課題であり、Apoptosis, Necrosis, ETosisなどその細胞や組織・臓器レベルでの詳細メカニズムは解明されつつあります。一方、個体レベルでの死のメカニズムは未だほとんどが謎に包まれています。そこで、我々は、個体の死が、単に従来の細胞死や臓器不全の蓄積・伝播によるのではなく、個体レベル特有のメカニズムが存在すると考えました。そして、この仮説を検証するために、動物種を超えた普遍的な(特に脊椎動物に共通の)個体レベルの生命機能や疾患に関わる遺伝子を同定するのに優れているゼブラフィッシュを用いて、細胞や組織・臓器レベルでの既知の死のメカニズムに囚われないBias-Freeのアプローチで個体レベルでの死の制御に関わる遺伝子をスクリーニングしました。その結果、個体レベル特有の「死」の制御に関わっている複数の候補遺伝子を同定しました。今後は、これらの遺伝子の機能がどのようなメカニズムで個体の死を制御しているかを明らかにしていく予定です。本研究は、生物学・医学における根幹的で未解決の問いである「個体死の制御機構」を、斬新な発想と独自の実験系で解決する提案であり、その成果は、生物学・医学の体系や方向を大きく変革・転換させ飛躍的に発展させる潜在性を有すると考えています。
D. 全身網羅的な多器官連関ネットワークの解明
従来の疾患研究は、少数の限定的な器官や組織に限定されていました。しかし、多くの疾患は、1つや数個の器官だけではなく、多数の器官に影響を及ぼすことが分かってきています。そこで我々は、全身を網羅する全ての器官同士の複雑なクロストークによって生体の恒常性が保たれており、例え最初に影響があったのは1つの器官だとしても、その影響は、ほぼ全ての器官に伝播し、器官間の複雑なフィードバックにより、シンプルな直線的で単一方向の因果関係は崩壊する、と考えました。
さらに、この全身を網羅する多器官同士の多次元的なクロストークネットワークの作動原理を理解し、介入することができれば、その複雑な性質のために有効な治療医薬品が無い「多器官連関性疾患」の検査・診断や治療が可能になるのではないか、と考えました。例えば、これまでは、疾患器官そのものを主に治療対象としていましたが、多器官連関ネットワークを理解することで、器官Aの不全を治療するために、別の器官Bを対象とした創薬が可能になるのです。
この目的を実現するため、まずは、様々なヒト疾患の発症から悪化までの病態動態を反映するマウスの各種病態モデルにおいて、全身を網羅する13~24種の器官での遺伝子発現の変化をゲノム網羅的に計測・解析し、その全身地図を作成しました。本研究では、心臓疾患、腎臓疾患、糖尿病、がん、若年性認知症それぞれの疾患ステージの初期から後期をカバーする地図を完成し、この地図を「全身網羅的多器官遺伝子発現地図(iOrgans Atlas)」と名付け、データベースとして公開しました(i-organs.atr.jp)。
この地図をインフォマティクスの各種ツールで解析することで、これまで知られていなかった数多くの多様な多器官連関ネットワークの候補の存在が見えてきました。また、全ての疾患モデルの皮膚において、数多くの遺伝子の発現が、疾患および疾患ステージ特異的に変動していることも明らかになりました。これは、皮膚が体の異常に敏感に反応するセンサーあるいはバロメーターとして働いている可能性を示唆しています。
我々は、特に腎臓疾患およびそれに付随する関連疾患における皮膚の反応に注目し、その詳細を解析したところ、骨の細胞がこれらの疾患のかなり初期の予兆を感知し、FGF23というホルモンを血中に分泌し、このホルモンが介在することで、皮膚における計25個の特異的な遺伝子の発現変動が起こることが明らかになりました。さらに、これらの遺伝子の皮膚での発現変動のパターンの違いから、何が原因で腎臓疾患が発症しているのかを明らかにできる可能性があることも分かりました。
成果論文
- S. Kozawa, R. Ueda, K. Urayama, F. Sagawa, S. Endo, K. Shizaki, H. Kurosu, G. Almeida, S.M. Hasan, K. Nakazato, S. Ozaki, Y. Yamashita, M. Kuro-o, T.N. Sato (2018). The body-wide transcriptome landscape of disease models. iScience (Cell Press), 2, 238-268.
E. 全身網羅的な多器官連関ネットワークの制御にかかる新規の器官特異的分泌因子の発見
研究成果Dから、様々な予想外の器官間クロストークが見えてきました。その成果から、全身網羅的な多器官連関ネットワークの制御にかかる新規の器官特異的分泌因子を発見しました。現在、この特定の器官からのみ分泌される因子がどのようなメカニズムで、全身網羅的に様々な器官の機能を制御しているのかを明らかにすべく研究を展開しています。さらに、この制御機構のヒトにおける疾患との関係を探るべく臨床研究も展開しています。
F. 全身網羅的な多器官連関ネットワークから発掘されたその他の予防・治療ターゲット
研究成果Dから、様々な予想外の器官間クロストークがあぶり出され、研究成果Eに示した成果以外にもいくつかの新規クロストークが見えてきました。現在、これらをターゲットとした治療薬開発のための適応症の絞り込み、および前臨床試験を産学連携の共同研究で実施しています。
G. 全身網羅的な多器官トランスクリプトームを活用したバーチャル創薬・バーチャル治験を可能にするAIシステム(humanized Mouse DataBase: hMDB)の構築
医薬品開発の現場では、前臨床・非臨床試験(培養細胞や動物での試験)で副作用の少ないことや効能を確認しますが、それらを検証できた被験薬の約90%が、その後、治験(ヒトにおける試験)で、副作用が生じる、または有効性が認められないなどの理由で開発中止になっています。開発中止による損失は1被験薬あたり150億〜2,000億円程度であるといわれており、昨今の医薬品の価格高騰の一因とされていることから、現在、医薬品開発のローリスク、ローコスト化が急務となっています。
治験で前臨床・非臨床試験と同じ結果を得られない主な原因は、細胞およびヒト以外の動物(マウス、ラット、ブタ、サルなど)とヒトとの”種差”に由来するため、これまでにも種差を解消するための手法が数多く開発されてきました。しかし、いずれも種差に由来するこの問題を根本的に解決するには至っていません。
我々は、2018年に全身網羅的な多臓器連関トランスクリプトーム地図(i−Organs Atlas)を完成させました。また、i−Organs Atlasと、新たに計測したマウスの全身網羅的な遺伝子発現パターン(トランスクリプトーム)とを照合することで、疾患の発症・重症化の仕組みや医薬品の作用を鮮明に映し出せることを発見しました。これらの背景のもと、本研究では、i−Organs Atlasを用いた医薬品の作用予測の実現を目的として、humanized Mouse DataBase(hMDB)というAIシステムを確立しました。
その結果、医薬品の構造やその標的に関係なく、その医薬品をマウスに投与し、全身網羅的な多臓器の遺伝子発現パターンを計測して計算機に入力することで、①副作用・有害事象5,519項目それぞれの発生有無およびその頻度、②世の中に知られている疾患11,312種類への効能の有無を予測できることが示されました。
また、性別、年齢層で層別化した各医薬品のヒトにおける副作用データを計算機に学習させることにより、性別、年齢層別に副作用・有害事象とその発生頻度を予測できることが示されました。〔層別化データを用いてhMDBで各層の予測を行う手法を「humanized Mouse DataBase, individualized(hMDB−i)」と命名しました。
効能の予測においては、hMDBに、各医薬品と、その投与により生じることが実社会(リアルワールド)で報告されている有害事象とをリンク予測(Link Prediction)で関連づけることにより(この手法を「hMDB/LP法」と命名しました。)、対象医薬品の既知の効能はもとより、これまで報告例のない効能を予測できることが示されました。これは、本手法がドラッグ・リポジショニングに活用できることを示唆しています。
hMDBによる副作用・有害事象の予測においては、AIの思考回路をのぞき見ることで、医薬品のどのような特徴をAIが捉えて予測を出力したのかを紐解くことができ、その特徴を解析することで、各医薬品の予測される副作用、有害事象発生の生物学的メカニズムを推論できることが示されました。
我々が開発したhMDBおよびhMDB/LP法は、予測能の比較において、医薬品開発で主流となっている予測システムの一つである「LINCS L1000」〔提供元:アメリカ国立衛生研究所(NIH)〕(http://lincsproject.org)など複数の既存AI予測システム(医薬品に付随する構造、遺伝子発現、標的、相互作用など多数の特徴をAIで解析して副作用や効能を予測するシステム)より優れた予測能を示しました。
今回、hMDB、hMDB/LP法がモダリティ・インディペンデントかつバイアス・フリーで医薬品の副作用・有害事象および効能を高精度に予測できることを実証しましたが、なぜその予測が可能なのかといった仕組みの理解は、まだ十分ではありません。
そこで我々は、全身網羅的な複数臓器の遺伝子発現パターンが、生体およびリアルワールドのさまざまな情報とリンクしており、この遺伝子発現パターンがこれら他の複雑系情報を映し出す「万華鏡」のような役目をしているとの仮説に基づき、その仕組みの解明に取り組んでいます。
成果論文
- S. Kozawa, F. Sagawa, S. Endo, G. M. De Almeida, Y. Mitsuishi, T. N.Sato (2020) Predicting Human Clinical Outcomes using Mouse Multi−Organ Transcriptome. iScience (Cell Press), 23, 100791.
H. 世の中に存在する全医薬品の全ドラッグリポジショニング標的辞書のAIによる作成
研究成果Gの結果から、hMDBは新薬候補のみならず既存薬の適応拡大(ドラッグリポジショニングなど)のための対象疾患選定にも活用できることを示しました。しかし、現在、世の中には約5,000の医薬品が既に存在しており、これら一つ一つをhMDBの予測システムで評価するためには、膨大な費用がかかります(1医薬品をマウスに投与し、24臓器のトランスクリムトームデータをとる必要があるため)。
そこで、この多臓器トランスクリムトームデータを介さないで、既存薬の既に存在するReal-World-Data (RWD)のみからそれぞれの適応拡大のための標的疾患の予測ができるAIシステムの研究開発を行いました。そして、そのシステム(Virtual Drug Repositioning: V-DR)をIn silicoのRetrospective blinded studyで評価し、その有効性を示すことができました。
I. より精度の高い医薬品の適応症の網羅的予測及び作用メカニズムを見える化できるAIシステムの開発
研究成果Hのシステムは既存のRWD(特に、有害事象発生頻度の情報)が無い医薬品には使えない、つまり、「新薬候補」には使えないという欠点があります。一方、研究成果GのhMDBは、新薬候補にも使えるのですが、さらなる予測精度の向上の可能性を秘めています。そこで、hMDBよりも予測精度のさらに高いAIシステムを確立することに成功しました。また、このシステムを使うと、被験物質が何故その適応疾患に効果を発するのかに関する分子細胞臓器レベルでのメカニズムの予測もできるようになりました。
J. バーチャル全身全細胞トランスクリプトーム地図の作成と活用
本プロジェクトのキーワードの「全身全臓器全細胞ライブ予測」に立脚し、プロジェクト開始当初の2013年から、全身全臓器全細胞ライブ予測の基盤技術となるツール・レファランスの一つとして、全身の全細胞における全遺伝子発現地図作りに役立つ技術の研究開発を行いました。それが、ゼブラフィッシュを用いてのsingle molecule FISHによる、生体内の一細胞レベルでの複数遺伝子発現の可視化でした。その結果は、2015年に論文(Oka & Sato, Sci Rep, 2015)として報告しました。しかし、Human Cell Atlasやその他関連技術の世界的な動向を踏まえ、もっと効率的に生体の全細胞網羅的遺伝子発現地図を作れる技術の研究開発をすることに方向転換しました。
そこで、我々が選択したのは、各臓器のRNA-seqデータを計算機上で、一細胞トランスクリプトームにDeconvolutionし、バーチャルなsingle-cell RNA-seqを作れる技術の研究開発です。様々な動物モデル(正常マウスや疾患モデルマウスなど)やヒトの膨大な臓器のRNA-seqデータはすでに存在するので、この技術ができれば、即座に全臓器のVirtual single-cell RNA-seq地図(V-scRNAseq Atlas)ができると考えました。そして、V-scRNAseq Atlasを完成させることに成功しました。
成果論文
- Y. Oka, T.N. Sato (2015) Whole-mount single molecule FISH method for zebrafish embryo. Scientific Reports, 5:8571.
K. その他
上記の主たるプロジェクト以外に、以下の成果論文も発表しました:
- Y. Sato, K. Yasuhara, J-i. Kikuchi, T.N. Sato (2013) Synthetic cell division system: Controlling equal vs. unequal divisions by design. Scientific Reports, 3: 3475.
- K. Okabe, S. Kobayashi, T. Yamada, T. Kurihara, I. Tai-Nagara, T. Miyamoto, Y.S. Mukouyama, T.N. Sato, T. Suda, M. Ema, Y. Kubota (2014) Neurons limit angiogenesis by titrating VEGF in retina. Cell, 159:584-96.
- L-J. Duan, S.J. Pan, T.N. Sato, G-H. Fong (2017) Retinal angiogenesis regulates astrocytic differentiation in neonatal mouse retinas by oxygen dependent mechanism. Scientific Reports, 7, 17808.
L. 完全独立型若手グループリーダーの成果論文
河岡グループ:
- Hojo MA, Masuda K, Hojo H, Nagahata Y, Yasuda K, Ohara D, Takeuchi Y, Hirota K, Suzuki Y, Kawamoto H, and Kawaoka S. Identification of a genomic enhancer that enforces proper apoptosis induction in thymic negative selection. Nature Communications 2019; 10: 2603.
- Enya S, Kawakami K, Suzuki Y, and Kawaoka S. A novel zebrafish intestinal tumor model reveals a role for cyp7a1-dependent tumor-liver crosstalk in causing adverse effects on the host. Disease Models & Mechanisms 2018; 11(8). pii: dmm032383.
- Hojo H, Enya S, Arai M, Suzuki Y, Nojiri T, Kangawa K, Koyama S, and Kawaoka S. Remote reprogramming of hepatic circadian transcriptome by breast cancer. Oncotarget 2017. 8:34128-34140