
総括責任者 堀越 正美
(東京大学 分子細胞生物学研究所 助教授)
研究期間:1997年10月~2002年9月
私達の身体は遺伝子DNAの遺伝情報に基づいて形作られますが、「細胞内において染色体DNAからどのようにして遺伝子が読み出されるか?」という機構に関しては多くの謎に包まれています。本プロジェクトでは、染色体構造をとっている遺伝子DNAの特定領域を選択して遺伝情報の読み出し(転写)を制御する蛋白質「ジーンセレクター」を単離し、その作用を解明する研究を行いました。
その結果、ヒストンのアセチル化修飾を介して染色体がグローバルに機能区分されるという、これまで全く考えられなかった遺伝子発現の制御機構を発見することに成功しました。また、ヒストンアセチル化酵素・脱アセチル化酵素に代表される正・逆反応酵素の活性中心付近に機能的な共通モチーフ構造を見出しました。さらに、「ジーンセレクター」の機能変異実験を通して、多細胞生物で知られているアポトーシス様の細胞死が単細胞生物においても存在することを発見しました。これらの成果は、いずれもオリジナルな概念に基づいて新仮説を構築することを目指したものであり、その結果、このような多くの新しい「芽」を多岐の研究分野に生みだすことに成功しました。
成果
染色体領域を機能区分する分子機構の発見
テロメア周辺領域において、ヒストンH4のアセチル化制御がSas2とSir2によって担われていることを発見した。さらに、Sas2がテロメアから離れたヒストンにアセチル基の目印を与え、Sir2がテロメア付近のヒストンからこの目印を取り除くことで染色体に「境目」をつけていることを世界に先駆けて明らかにした。
ヒストンアセチル化酵素と脱アセチル化酵素の共通モチーフ構造の発見
ジーンセレクター同士のアミノ酸配列を比較することにより、ジーンセレクターの分子内に新しいモチーフ構造を多数発見した。このうち、ヒストンアセチル化酵素Esa1およびヒストン脱アセチル化酵素Rpd3の両分子に共通する新規モチーフ構造(ERモチーフ)については、このモチーフが両酵素において機能的であることを明らかにした。
新規ヒストンシャペロンの発見
CCG1相互作用因子の1種として単離したCIA蛋白質が新規のヒストンシャペロンであることを見出した。また従来ヒストンシャペロンの活性に必要と考えられてきた分子内酸性ドメインについて、CIAでは試験管内・細胞内でともに不必要であることを見出した。さらに、転写開始制御の中心的因子であるTFIIDのサブユニットなどにヒストンシャペロン活性を見出した。
単細胞生物におけるアポトーシス様細胞死の発見
ヒストンシャペロンCIA1の欠損酵母において、核・クロマチンの断片化が観察され、さらに多細胞生物のアポトーシスの指標であるDNA 断裂、細胞膜反転、ミトコンドリアからのチトクロムc放出が検出された。また酵母因子UNI1を各種細胞で発現させるとアポトーシスの基準を多く満たす細胞死を起こした。これらの知見から、単細胞生物でも多細胞生物のアポトーシス様の細胞死機構が存在することが示唆された。
ヒストンの全染色体反応制御における機能解明
染色体の基本構造蛋白質であるヒストンに対してアミノ酸点変異株を包括的に樹立し、細胞増殖・転写・クロマチン構造変換などを制御するヒストンのアミノ酸を特定化することに成功した。さらに「ジーンセレクター」と相互作用するヒストンのアミノ酸残基を同定する戦略により、ヒストンの1アミノ酸相違を選択的に認識する薬理化合物を発見した。
各種「ジーンセレクター」のX線結晶構造の解明
ヒストンシャペロンCIAの立体構造解析を試み、出芽酵母CIA蛋白質の結晶作製とX線結晶回折パターンの取得に成功した。また蛋白質の機能を立体構造解析から推定することを試み、CCG1相互作用因子である新規蛋白質CIBのX線結晶構造を解明した。さらに、細胞周期制御因子の酵母ホモログの構造を明らかにすることに成功し、CDKによるRBリン酸化の特異性を合理的に説明する新知見を得た。
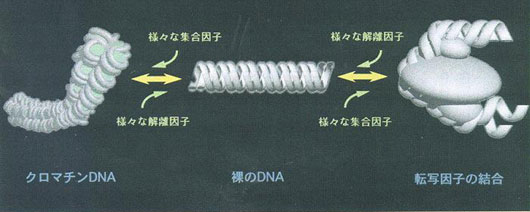
▲遺伝情報の読み出しと染色体DNAの構造変換に関するモデル