事業成果
研究データをひろく、つなげて、つかいやすく
生命科学データの利活用のための研究開発2025年度更新
 ライフサイエンスデータベース統合推進事業
ライフサイエンスデータベース統合推進事業
NBDC事業推進室
生命科学における新たな知識の創出を促進するため、国内外のさまざまな機関と連携し、生命科学データベースに関する中核的な研究データベースの整備と基盤的ウェブサービスの提供を通じたデータベースの統合化に取り組む。
事業概要
中核的な研究データベースの整備 (統合化推進プログラム)
「統合化推進プログラム」は、ライフサイエンスに関わる国内外のデータを統合的に扱うためのデータベース開発を目的とした競争的研究費制度。研究データ利活用のための情報環境を整備し、利用者の知識発見や課題解決、イノベーション創出への寄与および国際的なオープンサイエンスへの貢献を目指す。
現在、本格型として8件、育成型として6件、合計14件を支援中。
2024年度成果
空間トランスクリプトミクスデータベース「DeepSpaceDB」を公開 生体組織の複雑な内部構造や疾患メカニズムの解明への貢献に期待
京都大学 医生物学研究所 VANDENBON Alexis准教授らは、空間トランスクリプトミクスデータベース 「DeepSpaceDB」を2024年9月に公開した。
DeepSpaceDBではさまざまな生体組織の空間トランスクリプトミクスデータ(どの場所でどの遺伝子がどの程度発現しているか)を簡単に閲覧し、バイオインフォマティクスの専門家でなくても、容易にかつインタラクティブに解析できる。現在世界中で公開されているほぼ全ての空間トランスクリプトミクスデータを収載し、遺伝子発現プロファイルに基づく各スポットのクラスタリング、任意遺伝子の空間的発現分布、空間的に変化しているパスウエイ、発現遺伝子から予測された細胞の種類の空間的分布が表示でき、任意に選択したエリア間での遺伝子発現比較なども可能である。DeepSpaceDBを活用することで、組織切片のどの領域にどのような種類の細胞が存在するのか、腫瘍サンプルでどの辺りにどのような免疫細胞が浸潤してきているのかを見出すことができる。
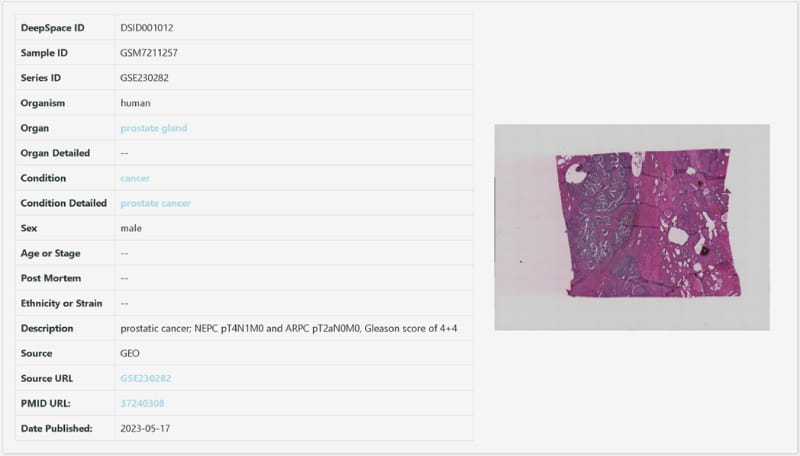
図1 組織サンプルの一例(DSID001012、ヒト前立腺がんの腫瘍切片)

図2 任意に選択したエリア間での遺伝子発現比較
set 1 (腫瘍周辺部のルミナル細胞)とset 2 (腫瘍から離れた位置のルミナル細胞)の領域で、発現に差のある遺伝子を抽出。

図3 HMOX1遺伝子の発現分布腫瘍周囲の組織において、線維化を示すマーカーであるHMOX1 (heme oxygenase 1)遺伝子の発現が高くなっていることが確認できる。
植物共発現データベース「ATTED-II」に新たな解析ツールを実装 モデル植物の遺伝子共発現の知見を非モデル植物の研究に活用
東北大学 大学院情報科学研究科 大林 武 教授らは、2024年9月、植物の遺伝子共発現データベース「ATTED-II」に、遺伝子の共発現関係を閲覧・解析するための新たな機能を追加した。
ATTED-Ⅱはシロイヌナズナなどのモデル植物を中心に、主な植物種のトランスクリプトームデータを大規模に解析・収載した、特定の条件や環境で発現する(共発現遺伝子)を探索できるデータベースである。共発現遺伝子は一連の生理的機能を担っていることが多く、機能のまとまりや遺伝子の制御関係の理解に役立つが、モデル植物における遺伝子の共発現は、未知の部分が多い穀物や野菜などの「非モデル植物」での遺伝子の機能を知るためにも有用である。
新たに実装された「PC View」では、着目する2つの遺伝子が共発現する条件や環境を調べることができ、また、「CoexViewer」では着目する2つの遺伝子の発現の相関関係を確認できるようになった。これらのツールを使うことで、共発現遺伝子が担う生理的機能やその制御関係の考察が容易になった。

図4 「PC View」の画面共発現関係にある遺伝子の発現条件の PC (主成分) を説明するページ。ここでは、(1) サンプルの発現量データに対するPC分析結果のプロット図、(2) 第1から第10までの各PCに関与する代表的な6つのサンプル (負荷量 (PCA loading) が最も高い3つのサンプル (赤字) と最も低い3つのサンプル (緑字)) が含まれるstudy10件、(3) 発現量が正に相関している上位100遺伝子のリスト、(4) 発現量が負に相関している上位100遺伝子のリストが示される。
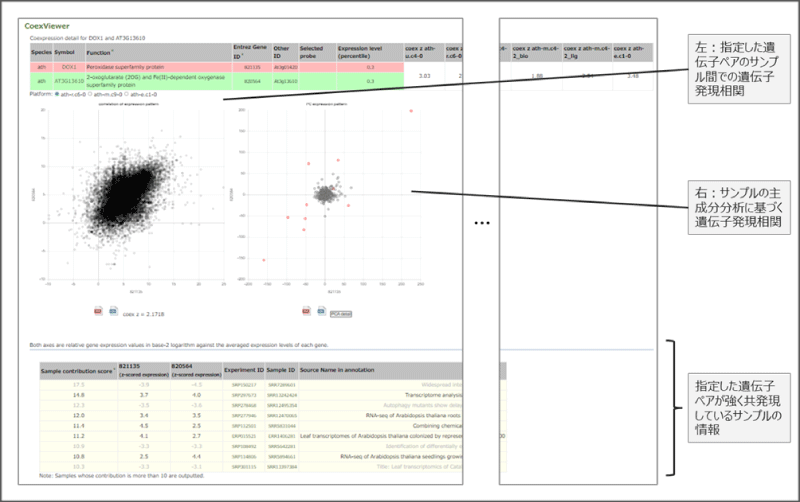
図5 「CoexViewer」の画面指定した遺伝子ペアの遺伝子発現相関を表示する。本図は、シロイヌナズナのPeroxidase superfamily proteinと2-oxoglutarate and Fe(II)-dependent oxygenase superfamily proteinの結果。サンプル間での遺伝子発現相関と、サンプルの主成分分析に基づく遺伝子発現相関から、共発現の背景にある生物学的な意味を理解することができる。
日本人ゲノム研究基盤の新たな一歩:「TogoVar」への大規模プロジェクト由来データ拡充と新ビューア実装
日本人ゲノム多様性統合データベース「TogoVar」は、2024年、新たにバイオバンク・ジャパン(BBJ)とナショナルセンター・バイオバンクネットワーク(NCBN)のデータを収載、タンパク質立体構造ビュータを実装し、ゲノム創薬等に資するデータ基盤として拡張された。
TogoVarはライフサイエンス統合データベースセンター (DBCLS)が開発する日本人ゲノム多様性統合データベース。2018年の公開以降、国内外の多数のプロジェクトと連携してバリアント(ゲノム配列の個人間の違い)情報を集約し、データ利活用を促進してきた。
2024年6月新規収載のBBJおよびNCBNデータには同名の国内研究プロジェクトによって得られた日本人集団の情報が多数含まれる。既収載の国際的なデータセットと比較することで、日本人集団の特徴などをさらに高精度に検証できるようになった。
12月実装のビューアでは、ミスセンスバリアントのタンパク質の立体構造上の位置が確認できる。ゲノム情報とタンパク質情報の統合により、バリアントのタンパク質の機能や疾患への影響などの推測が容易になった。
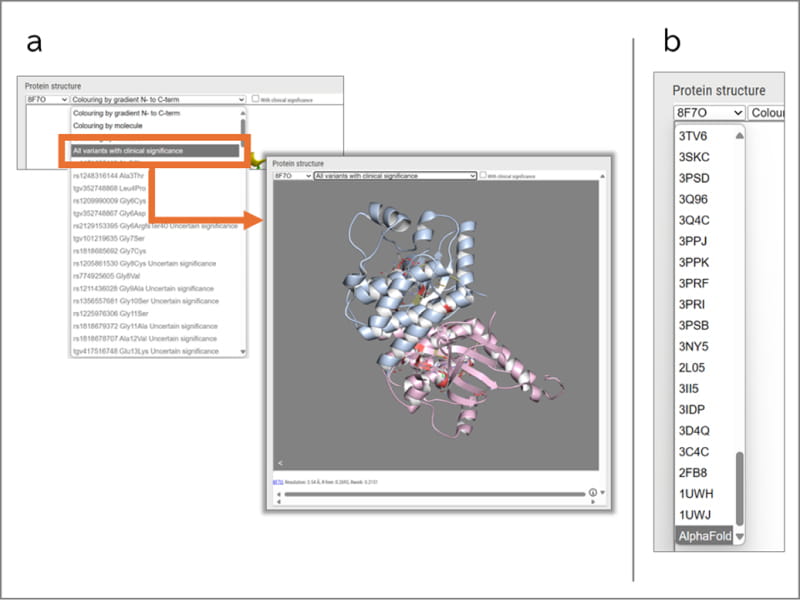
図6 2024年12月実装の「Protein structure」ビューア(例:BRAF:p.V600E)。a) ビューアの右側のプルダウンメニューから「All variant with clinical significance」を選択すると、臨床情報のあるバリアントの位置が赤く強調表示される。
b) 同ビューア内の左側のプルダウンメニューでは、複数の構造データがPDBへ登録されている場合にどれを表示するか選択できる。AlphaFoldによる予測構造データも選択可能。PDBの実測データは大阪大学のPDBjから、AlphaFoldの予測データはEMBL-EBIのAlphaFold DBから取得している。
なお、図で例示したBRAFはプロテインキナーゼの一種。活性型RASと結合して活性化し、MEKのリン酸化を通じて細胞増殖を促進する。いろいろな種類のがんでBRAFの点変異が知られ、阻害薬も開発されている。