ポイント
- TogoVar(トーゴーバー:日本人ゲノム多様性統合データベース)では、ゲノム配列の個人による違い(バリアント)に関するさまざまな条件を用いて、国内外のデータベースや文献情報などのワンストップ検索を可能にした。
- 検索対象には、NBDCヒトデータベースに登録された日本人のゲノムデータから集計した大規模なバリアントの頻度情報が含まれており、この頻度情報のデータセットもTogoVarから公開する。
- 今後、さらなる情報の充実を図り、日本人を対象とした個別化医療(高精度医療)などのゲノム医科学に寄与する日本人ゲノム情報基盤を目指す。
JST(理事長 濵口 道成)と情報・システム研究機構(機構長 藤井 良一)は、日本人ゲノム配列の個人による違い(バリアント)とそれに関係する疾患情報などを収集・整理したデータベース「TogoVar」を構築し、平成30年6月7日より無料公開します。
薬の効き方や疾患のかかりやすさ、お酒を飲むと顔が赤くなるといった体質などの「表現型」は、遺伝子のバリアントと関係しています。表現型とバリアントの関係を発見するためには、対象とする集団に存在するバリアントの割合(頻度)の情報が必要であり、多くのデータを活用できることが成功の鍵となります。すでに海外では大規模な個人ゲノムデータを集約したバリアントの頻度情報が公開され、広く利用されています。一方、日本国内では、これまでさまざまな研究プロジェクトごとに公開されてきたため、プロジェクトを越えて横断的にバリアントの頻度情報を活用できるようにすることが課題でした。
この課題を解決するために、TogoVarでは、各プロジェクトで生産された個人ゲノムを集計したバリアントの頻度情報や文献情報などを収集・整理し、さまざまな条件(バリアントのヒトゲノム上の位置、種類など)を用いて、ワンストップで検索する機能を提供します。これと同時に、これまでNBDCヒトデータベース注1)に登録された日本人のゲノムデータから大規模なバリアントの頻度情報を集計して検索対象とするとともに、そのデータセットをTogoVarで公開しました。
今後、TogoVarは、遺伝カウンセリングなど日本人を対象にした個別化医療(高精度医療)に向けたゲノム医科学の発展に寄与する日本人ゲノム情報基盤となることを目指し、バリアントに付随する遺伝子発現データなどの研究者に有用な情報を追加するとともに、NBDCヒトデータベースによりバリアントの頻度情報をさらに充実させていきます。
TogoVar URL:https://togovar.biosciencedbc.jp
本データベースは、科学技術振興機構(JST) バイオサイエンスデータベースセンター(NBDC)のライフサイエンスデータベース統合推進事業の一環として、NBDCと情報・システム研究機構 データサイエンス共同利用基盤施設 ライフサイエンス統合データベースセンター(DBCLS)との共同研究により、開発されました。
<ゲノム多様性統合データベースの背景>
薬の効き方や疾患のかかりやすさ、お酒を飲むと顔が赤くなるといった体質などを「表現型」といい、表現型はゲノム配列の個人による違い(バリアント)と関係しています。表現型とバリアントの関係を発見するためには、表現型の有無により分けたそれぞれの集団におけるバリアントの頻度情報が必要です。日本人は白人集団に比べて耳垢が乾いている人の割合が多い、また、それほど太っていなくても糖尿病を発症する人が多いなど、集団によるバリアント頻度の違いが表現型の割合の違いに反映されることがあります。
これまで日本では複数の公的研究プロジェクトにおいてそれぞれヒトゲノムのデータベースが構築され、集計情報などが公開されていますが、これらは研究プロジェクトごとの形式によるデータセットからの情報であることがほとんどです。それぞれの研究プロジェクトにおいて構築され、乱立するデータベースを1ヵ所に集約し、横断的に整理統合することで、大規模集団でのバリアントの頻度情報を低コストで簡単に多くの研究者が利用できるようになるため、このように統合されたデータベースの価値は飛躍的に高いものとなります。
すでに海外では、大規模なバリアントの統合データベースとしてdbSNP注2)やExAC、gnomAD注3)などがあり、10万人規模の個人ごとのゲノムデータを集約したバリアントの頻度情報が公開され、広く利用されています。
日本人集団については、このように研究プロジェクト横断的に統合した大規模集団でのバリアントの頻度情報がなく、日本人を対象にした個別化医療(高精度医療)に向けたゲノム医科学を推進する上での大きな課題でした。そこで、日本版のExAC、gnomADと呼べる、日本人ゲノム情報基盤の構築が求められていました。
<これまでのヒトデータベース構築の経緯>
近年、公的資金を投じた研究で生産されるさまざまなデータが公的データベースへ登録されることで、データの利活用が促進されています。
このような「オープンサイエンス」の取り組みの1つとして、JST バイオサイエンスデータベースセンター(NBDC)は、個人情報の保護に配慮しつつヒトに関するさまざまなデータを共有するための公的なプラットフォーム「NBDCヒトデータベース」を構築し、2013年10月より運用を開始しました。NBDCヒトデータベースの運用は国立遺伝学研究所 DNA Data Bank of Japan(DDBJ)と協力して行っており、個人ごとのゲノムデータやさまざまな表現型情報は、DDBJが構築した「Japanese Genotype-phenotype Archive(JGA)注1)」に格納され、共有されています。
NBDCヒトデータベースは、学術や公衆衛生の向上に資する研究であれば国内の研究機関のみならず民間企業や海外の機関にもデータを共有することで、データの利活用を促進してきました。
この度運用を開始する日本人ゲノム多様性統合データベース「TogoVar」は、これまで構築してきた仕組みを生かし、発展させたものです。
<日本人ゲノム多様性統合データベース「TogoVar」の内容>
1.散在するデータを整理統合して、ワンストップでわかりやすく提供します
個々のバリアントが表現型に及ぼす影響を解釈するには、さまざまな知見や情報から統合的に判断する必要があります。そのため、東北メディカル・メガバンク機構や京都大学などから公開されているバリアントや遺伝子発現に関する情報、ClinVar注4)のようなバリアントと疾患の関係に関する情報、日々更新される文献情報など、多くのデータベースをまたぐ情報を継続的に収集し続けなければなりません。TogoVarではResource Description Framework(RDF)注5)を用いて、多種多様なデータベースに散在して収録されてきた遺伝子型や表現型に関連する情報を整理統合し、ワンストップでわかりやすく提供します(図1、2)(表1)。これらを用いて、国内外のデータベースを比較することで、特定のバリアントに関する日本人と日本人以外の集団との出現頻度の比較などを可能にしました。
2.日本人におけるバリアントの頻度情報を提供します
TogoVarでは、NBDCヒトデータベースに登録されている個人ごとのゲノムデータを集約して得られた「日本人におけるバリアントの頻度情報」を提供します(図3)。今回日本人125人分の全エクソン注6)データから集約した約1,300万ヵ所のバリアントと、183,884人分の既知SNP注7)データから集約した約200万ヵ所のバリアントを収録しました。これらを用いて、例えば、難病研究において、原因となるバリアント候補の絞り込みや多因子疾患の解析が可能になります。
3.既存データベースでIDを持たなかったバリアントにもIDを付与しています
TogoVarでは、収録するすべてのバリアントにIDを付与します(図1)。これまでdbSNPなど既存の国際的なデータベースに収録されていなかった多数のバリアントについても今回初めてIDを付与し、合計6,700万個以上の全バリアントに独自IDを付与しました。これにより、これまでIDが付与されていなかったバリアントデータも見つけやすくなり、学術論文などで容易に引用できるようになりました。
<今後の展開>
1.日本人ゲノム情報基盤としてゲノム医科学研究や先端医療への貢献を目指します
研究者はTogoVarを活用することで、過去の複数の研究プロジェクトにおいて取得された日本人のゲノムデータを効率よく収集し、自身の研究に用いることが可能になり、疾患などに関連した新たなバリアントの検出につながると期待できます。また、検出されたバリアントの解釈に必要となる既知の情報や日本人以外の集団における頻度情報などもワンストップで取得できることにより、ゲノム医科学研究における情報収集の迅速化や研究の効率化を支援します。 さらに、遺伝カウンセリングなどの個別化医療(高精度医療)を提供する際の参考情報としての利用など、先端医療へ貢献することを目指します。
2.個人ゲノムデータをさらに追加し、バリアントの頻度情報の品質を向上させます
近日中にNBDCヒトデータベースに1,026人分の全ゲノムデータが追加され、TogoVarにも反映されます。NBDCヒトデータベースへの個人ゲノムデータの登録が増えることで、より大規模な日本人集団における頻度情報に更新され、TogoVar収録データの品質がさらに向上していきます。今後も随時収録可能なデータを追加していく予定です。
<参考図>
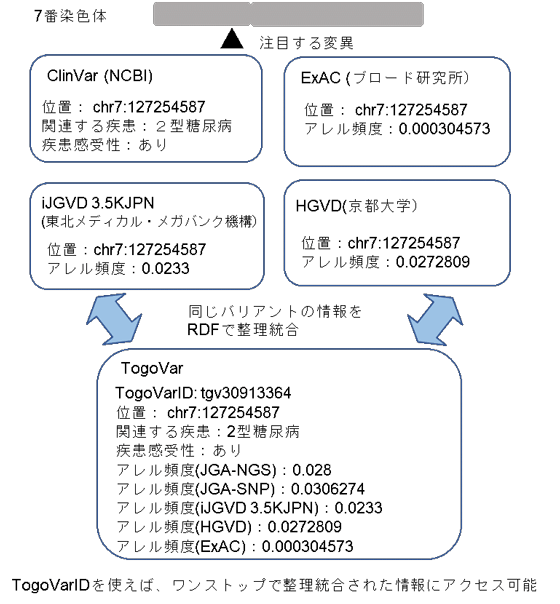
図1 ID付与による情報の整理統合とワンストップ検索
整理統合した全バリアントにTogoVarIDを付与(日本版dbSNP/dbVar)。散在する、バリアントの解釈に必要な情報も整理統合した。
バリアントの位置情報は各データベースが参照するゲノム配列のバージョンによって異なることがある。また、バリアントに関連する情報はデータベースごとの目的によって違いが生じる。これらの違いを吸収し、複数のデータベースを渡り歩くことなく、関連する情報をワンストップで取得できる。
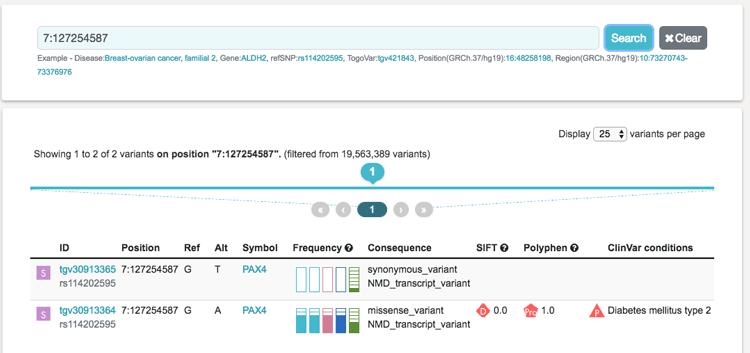
図2 TogoVarのワンストップ検索結果の例
ある患者群において、7番染色体の127254587番目の塩基がGからAに変わるバリアントが多く発見されたので、ゲノム上のバリアントの位置を条件に既知の情報を検索した例。ClinVarでは2型糖尿病との関連が示唆されていることに加え、日本人集団(JGA、3.5KJPN、HGVD)での頻度が、欧州人を主とするExACでの頻度よりも100倍程度高いことがわかる。
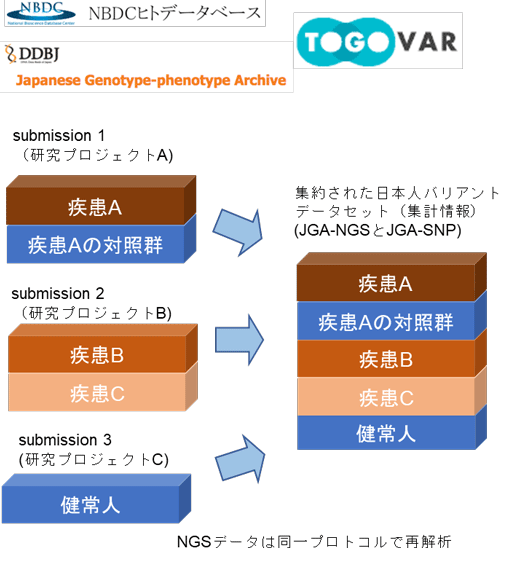
図3 日本人約18万人のバリアントの頻度情報の作成
TogoVarでは、NBDCヒトデータベース/JGAに登録されている日本人ゲノムデータを集約し、そのバリアントの頻度情報を公開する。
| データセット |
サンプル
サイズ |
バリアント
部位数 |
対象 |
バリアントの種類 |
| 一塩基置換 |
挿入・欠失 |
| JGA-NGS |
125 |
12,980,041 |
全エクソン |
✔︎ |
✔︎ |
| JGA-SNP |
183,884 |
1,958,606 |
既知SNP |
✔︎ |
|
| 3.5KJPN |
3,554 |
50,099,977 |
全ゲノム |
✔︎ |
|
| HGVD Ver.2.30 |
1,208 |
501,556 |
全エクソン |
✔︎ |
✔︎ |
表1 収録した日本人バリアントデータセット
東北メディカル・メガバンク機構が公開する3.5KJPNと京都大学が公開するHGVD Ver.2.30のサンプルサイズとバリアント部位数は、データを各公開ウェブサイトからダウンロードしNBDCが計数した。
<用語解説>
- 注1)NBDCヒトデータベース/Japanese Genotype-phenotype Archive(JGA)
- NBDCとDDBJが共同運営する、ヒト由来試料からのゲノムデータなどを共有するための公的リポジトリ。個人ごとのゲノムデータやさまざまな表現型情報について、データの提供および利用に関する申請はNBDCヒトデータベースを通じて行われ、登録されるデータはDDBJ内のJGAデータベースに格納されている。データ利用申請が承認されれば、TogoVarで集計される前の個人ごとのゲノムデータも利用できる。
- NBDCヒトデータベース URL:https://humandbs.biosciencedbc.jp/
- JGA URL:https://www.ddbj.nig.ac.jp/jga/index.html
- 注2)dbSNP
- 米国国立衛生研究所 国立生物工学情報センターが運営する1~数塩基の置換、挿入/欠失、反復などのゲノム多型情報からなるデータベース。
- 注3)ExAC(Exome Aggregation Consortium)、gnomAD(Genome Aggregation Database)
- 米国ブロード研究所が運営する世界中の大規模配列解析プロジェクトから収集した個人ごとのゲノムデータからバリアントを検出して集約したデータベース。ExACには約6万人のエクソーム注6)解析によって検出したバリアントが、その後継であるgnomADには約12万人のエクソーム解析と約1.5万人の全ゲノム解析から検出したバリアントがそれぞれ収録されている。
- ExAC URL:http://exac.broadinstitute.org/
- gnomAD URL:http://gnomad.broadinstitute.org/
- 注4)ClinVar
- 米国国立衛生研究所 国立生物工学情報センターが運営するバリアントの臨床的意義(clinical significance)を整理したデータベース。
- ClinVar URL:https://www.ncbi.nlm.nih.gov/clinvar/
- 注5)RDF(Resource Description Framework)
- インターネット上にあふれる情報を活用するには、コンピューターで自動的に処理し、高度に利用する技術が必要である。そこで、インターネットの国際標準化団体であるワールド・ワイド・ウェブ・コンソーシアムは、インターネット上の情報をコンピューターでより処理しやすいものにするための国際的な標準形式としてRDF形式を提案している。RDF形式で記述されたデータは、コンピューターが自動的に処理し、相互運用可能となる。研究者は多種多様なデータを連携させて利用できるようになる。
- 注6)エクソン、エクソーム
- ゲノム配列はたんぱく質を生成する元の情報となる領域と使用されない領域に分かれており、前者の領域をエクソン(exon)と呼ぶ。ゲノム中のすべてのエクソンの総体をエクソーム(exome)と呼ぶ。
- 注7)SNP(Single Nucleotide Polymorphism)
- ゲノム上のバリアントのうち、一定以上の頻度で存在する一塩基置換のこと。
<本成果が貢献しうる持続可能な開発目標(SDGs)>
<お問い合わせ先>
<TogoVarデータベースに関すること>
三橋 信孝(ミツハシ ノブタカ)
科学技術振興機構 バイオサイエンスデータベースセンター(NBDC)
〒102-8666 東京都千代田区四番町5番地3
Tel:03-5214-8491 Fax:03-5214-8470
E-mail:
片山 俊明(カタヤマ トシアキ)、川島 秀一(カワシマ シュウイチ)
情報・システム研究機構 データサイエンス共同利用基盤施設
ライフサイエンス統合データベースセンター(DBCLS)
〒277-0871 千葉県柏市若柴178-4-4 東京大学柏の葉キャンパス駅前サテライト6階
Tel:04-7135-5508 Fax:04-7135-5534
E-mail:
<NBDC事業に関すること>
舘澤 博子(タテサワ ヒロコ)
科学技術振興機構 バイオサイエンスデータベースセンター(NBDC)
〒102-8666 東京都千代田区四番町5番地3
Tel:03-5214-8491 Fax:03-5214-8470
E-mail:
<報道担当>
科学技術振興機構 広報課
〒102-8666 東京都千代田区四番町5番地3
Tel:03-5214-8404 Fax:03-5214-8432
E-mail:
情報・システム研究機構 データサイエンス共同利用基盤施設
ライフサイエンス統合データベースセンター(DBCLS)
広報担当 箕輪 真理(ミノワ マリ)
〒277-0871 千葉県柏市若柴178-4-4 東京大学柏の葉キャンパス駅前サテライト6階
Tel:04-7135-5508 Fax:04-7135-5534
E-mail: