ポイント
- 脊椎動物のDNA配列はいまだ完全には解読されておらず、特にセントロメア領域は未解読の部分が多く従来のDNA解読技術では困難であった。
- DNA解読技術の改良により、脊椎動物のモデル生物であるメダカのセントロメア配列の約10%の解読に成功した。
- セントロメア配列のCpGメチル化状態の解析技術を開発し、染色体中心部分に存在するセントロメア配列の進化速度が速いことが明らかとなり、種分化に関わっている可能性のあることが分かった。
JST 戦略的創造研究推進事業において、東京大学の森下 真一 教授らと武田 洋幸 教授らは、長いDNA断片の解読技術を改良し、脊椎動物のモデル生物であるメダカのセントロメアDNA配列の約10%(約100万塩基)を解読することに成功しました。
21世紀に入りヒトをはじめとする哺乳類を含む、脊椎動物のDNA配列の解読が進みましたが、完全には解読されておらず、いまだに脊椎動物のDNA配列には数百から数十万の解読できていない領域が残されています。とりわけ、セントロメア注1)と呼ばれる長い領域は未解読のままでした。
本研究グループは、数万の塩基のDNA断片を解読する装置を活用し、DNA断片の解読を行いましたが、1分子のDNAを直接観測するためノイズが入るといった難点がありました。今回、ノイズを取り除くために複数の補完的な手法で補正した結果、脊椎動物のモデル生物であるメダカを対象に、セントロメアのDNA配列の約10%(約100万塩基)を解読することに成功しました。また、セントロメア配列のCpGメチル化注2)状態の解析技術についても開発し、染色体の中心に近いほど塩基変異が有意に蓄積されることを明らかにしました。ヒトゲノムの約1~2%はセントロメア配列が占めると推定されていますが未解明であり、今回のメダカゲノムの研究はその解明への手がかりにもなると期待されます。
本研究成果は、2017年11月28日(英国時間午前10時)発行の国際科学誌「Nature Communications」に掲載されます。
本成果は、以下の事業・研究領域・研究課題によって得られました。
戦略的創造研究推進事業 チーム型研究(CREST)
| 研究領域 |
「生命動態の理解と制御のための基盤技術の創出」
(研究総括:山本 雅 沖縄科学技術大学院大学 教授) |
| 研究課題名 |
「DNA3次元クロマチン動態の理解と予測」 |
| 研究代表者 |
武田 洋幸(東京大学 教授) |
| 研究期間 |
平成25年10月~平成31年3月 |
JSTはこの領域で、生命現象の統合的理解や安全で有効性の高い治療の実現などに向けたin silico/in vitroでの細胞動態の再現化による細胞と細胞集団を自在に操る技術体系を創出します。上記研究課題では、発生とゲノムの研究に有利なメダカをモデルとして、さまざまな分化段階の細胞からエピジェネティックコードおよびDNA接触情報を収集して、クロマチン動態を説明するロジックと数理モデルを構築します。
<研究の背景と経緯>
21世紀に入り、ヒトをはじめとする哺乳類を含む、脊椎動物のDNA配列の解読が進みましたが、そのすべてが完全に解読されたわけではありません。脊椎動物のDNA配列には、数百から数十万の解読が困難な領域が残されており、特にセントロメアと呼ばれる長い領域は多くが未解読でした。とりわけヒトゲノムのうち24本の染色体のセントロメアDNA配列の解読は、従来のDNA解読技術では困難でした。
<研究の内容>
セントロメア配列の解読を困難にしている理由は、ある単位配列が多数繰り返されるためです。例えば、ヒトゲノムの各染色体のセントロメアでは、171塩基のαサテライト配列注3)が何千回から何万回も繰り返されています。これらの配列は完全に同一ではなく、塩基が変異してある程度個性を持っていることがわかっています。これは、数百万匹の蟻を撮影した写真から作ったジグソーパズルを解くのに似ており、パズルのピースが大きいほど元の写真を再現しやすくなります。つまり、解読できる断片が長ければ長いほど正確に解読できるということです。
本研究グループは、非常に長い断片化したDNA(数万の塩基長)を解読可能な装置(Pacific Biosciences社)を使用し解読に取り組みました。この技術は独創的ですが、1分子のDNAを直接観測するため、どうしてもノイズが入ってしまいます。蟻の画像の例でいうと、動き回る蟻にピントを合わせられず画像がぼけてしまうことに似ています。そこで、ノイズを除くため複数の補完的な方法で補正をしました。その結果、脊椎動物のモデル生物であるメダカを対象に、セントロメアのDNA配列の約10%(約100万塩基)を解読することに成功しました。
次に、セントロメアのDNA配列がどのように進化してきたかを解明するために、種分化の過程にある3つの近交系メダカを分析しました(図1a)。2つは南日本由来および北日本由来で、日本列島の分水嶺が生まれた約1800万年前に分岐したと考えられています。他の1つは約2500万年前に分岐した韓国由来です。分析の結果、セントロメアの位置は3つの近交系でおおむね保存されていました(図1b)。より古い3.2~3.5億年前の真骨魚類の全ゲノム重複注4)のときにコピーされた染色体の組の中でも、4つの組はその後もセントロメアの位置を保存しています。セントロメア位置が保存される傾向の意味を考えることは今後の課題です(図2a)。
また、塩基レベルでも特徴的な性質が分かりました。セントロメアの塩基変異速度は他の領域より速く、種分化に関わるという説がありますが、詳細な分析は行われていませんでした。例えばセントロメアは染色体の中心だけでなく末端にも存在しますが、その存在位置が種分化と関係するかどうかは未解明でした。分析した結果、位置に依存して進化速度は異なり、セントロメアの中心に近いほどその塩基は有意に変化しやすいことが明らかになり(図2b)、セントロメアの位置が種分化に関わる可能性が示唆されました。
本研究では、セントロメアのDNA配列を解読し、さらにCpGメチル化状態を高解像度で分析する技術を研究開発しました。CpGはゲノム全体で1~3割は低メチル化状態にありますが、セントロメアにおいても同様であることが明らかとなりました。低メチル化したセントロメア領域に共通する顕著な塩基的特徴は見つからず、むしろ染色体ごとに低メチル化したセントロメア領域は個別の塩基変異を蓄積していることが分かりました。このような染色体内の独立した進化も種分化に関わっている可能性があると考えています。
<今後の展開>
ヒトゲノムのセントロメアDNA配列を解読し、その生理的な意義を解明することは、生命科学の中でも大きな研究課題の1つです。ヒトゲノムの約1~2%はセントロメア配列が占めていると考えられていますが未解明です。今回のメダカゲノムの研究は、その解明への手がかりになると期待されています。
<参考図>
今回の成果の概念図
性決定遺伝子および発生生物学などの研究に1世紀近く役立ってきたメダカが、セントロメアDNA配列の生物学的な意義の解明のためにも重要なモデル生物になると期待されている。
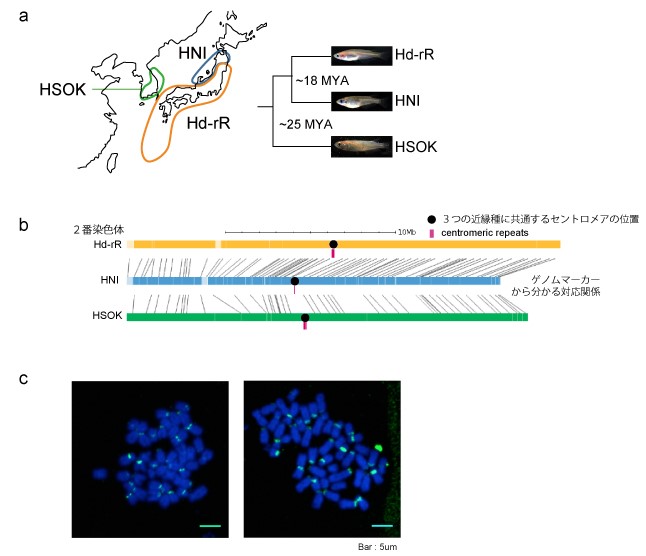
図1
- (a)種分化の過程にある3つの近交系メダカの地理的由来と進化系統樹。MYAは百万年前を意味する。
- (b)3つの近交系メダカの2番染色体でのセントロメア位置が保存されている。●はセントロメアの位置。
- (c)セントロメアを蛍光色素で染色した顕微鏡写真。
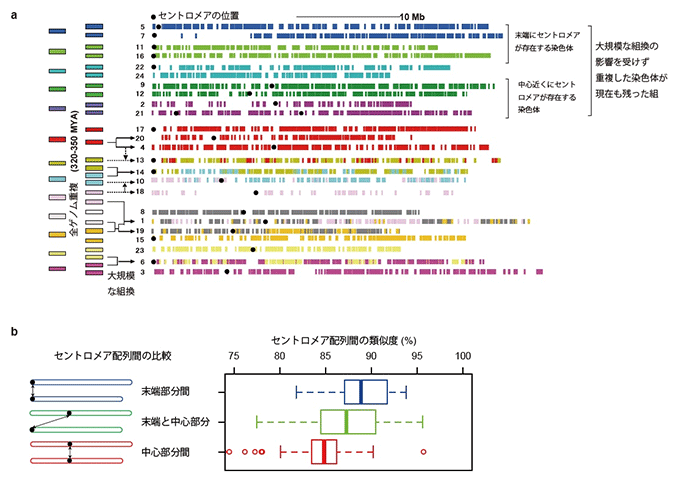
図2
- (a)3.2〜3.5億年前の真骨魚類の全ゲノム重複のときに重複した染色体の中で、染色体間の大規模な変化が起こらなかった5つの染色体の組の内、4つ染色体の組において、セントロメアの位置は保存されている。●がセントロメアの位置を示す。番号は染色体番号を示し、染色体中の空白は全ゲノム重複前の領域との対応情報が不足している領域を示す。
- (b)セントロメアは染色体の中心だけでなく、末端部分(acrocentric)にも存在するが、中心に近いほど塩基の変異が有意に蓄積されやすい。
<用語解説>
- 注1) セントロメア
- 染色体の短腕と長腕が交わる位置。染色体の中心に位置することが多いが、末端に存在する場合もある。ヒトゲノムの場合は171塩基前後のαサテライト配列が数千回から数万回繰り返していると考えられているが、未解明である。脊椎動物での繰返し配列の基本単位は種によって異なり多様であると考えられるが、未解明である。他のDNA領域に比べるとセントロメアの進化速度は非常に速く、5千万年以上前に分岐した生物種のセントロメア配列を比較すると、その相同性は25%まで落ちることが報告されている。
- 注2) CpGメチル化
- DNA塩基のシトシンのメチル化とは、5位の炭素原子にメチル基が付加する反応である。脊椎動物ではシトシンのメチル化の殆どがCpG部位で起こるため、特にCpGメチル化と呼ばれる。CpGメチル化は発生に必須であり、X染色体の不活性化、トランスポゾンの抑制、遺伝子発現の制御、ゲノム刷り込みなどのさまざまな機能にも関与する。セントロメア領域に代表される繰り返し配列でCpGメチル化を観測することはこれまで難しかった。
- 注3) αサテライト配列
- ヒトゲノムのセントロメア部分を構成する171塩基前後の基本単位。数千回から数万回繰り返していると考えられている。
- 注4) 全ゲノム重複
- ゲノム全体が重複する現象。例えば脊椎動物が分岐した初期のころ、全ゲノム重複が2回起こり、各遺伝子を4倍に増やし、脊椎動物の繁栄をもたらしたと考えられている。4倍に重複された個々の遺伝子は、進化の過程で新しい機能を獲得することもあり、一方で不要になる場合(偽遺伝子化)もある。哺乳類ゲノムではその痕跡が残り、1つの遺伝子に対して類似度が有意に高い配列が4つ見つかる場合が多い。脊椎動物内では、3.2〜3.5億年前に真骨魚類の祖先でさらにもう1回全ゲノム重複が起こっている。
<論文情報>
| タイトル |
“Centromere evolution and CpG methylation during vertebrate speciation”
(種分化過程での、セントロメア領域の進化とCpGメチル化) |
| 著者名 |
Ichikawa K, Tomioka S, Suzuki Y, Nakamura R, Doi K, Yoshimura J, Kumagai M, Inoue Y, Uchida Y, Irie N, Takeda H, Morishita S. |
| 掲載誌 |
Nature Communications |
| doi |
10.1038/s41467-017-01982-7 |
<お問い合わせ先>
<研究に関すること>
森下 真一(モリシタ シンイチ)
東京大学 大学院新領域創成科学研究科 教授
〒277-8562 千葉県柏市柏の葉5-1-5
Tel:04-7136-3985 Fax:04-7136-3977
E-mail:
武田 洋幸(タケダ ヒロユキ)
東京大学 大学院理学系研究科 教授
Tel:03-5841-4431 Fax:03-5841-4993
〒113-0033
東京都文京区本郷7-3-1
E-mail:
<JST事業に関すること>
川口 哲(カワグチ テツ)
科学技術振興機構 戦略研究推進部
〒102-0076 東京都千代田区五番町7 K’s五番町
Tel:03-3512-3524 Fax:03-3222-2064
E-mail:
<報道担当>
科学技術振興機構 広報課
〒102-8666 東京都千代田区四番町5番地3
Tel:03-5214-8404 Fax:03-5214-8432
E-mail: