理化学研究所(理研) 生命システム研究センター オミックス動態研究ユニットの城口 克之 ユニットリーダーらの共同研究チーム※1)は、従来の次世代シークエンサー注1)を用いた「DNA分子バーコード法注2)」を改良し、1万個以上の核酸分子(DNAおよびRNA)を正確に数える手法を開発したうえで、複数の試料由来の結果が混ざる問題も解決しました。
次世代シークエンサーは、一度に多数の核酸の配列を決定できます。城口ユニットリーダーらは2012年に、次世代シークエンサーを利用したDNA分子バーコード法を開発しました※2)。この方法では、それぞれ異なる配列を持つ短いDNA分子(DNA分子バーコード)を、目的の核酸分子の1つ1つに付加することで、核酸分子の数を数えることができます。以来、遺伝子発現量の解析などに応用され、多くの研究者や企業が利用し、かつ手法も改良されてきました。しかし、従来のDNA分子バーコード法では、次世代シークエンサーの配列決定でのエラーなどが原因で、100個程度の核酸分子までしか正確に数えられないという課題がありました。
今回、共同研究チームは、DNA分子バーコード法を改良するとともにDNAバーコードの長さをコンピュータ上で変えるなどの新たな配列情報解析法をおよび1万個以上の核酸分子を正確にデジタル計数注3)できる手法を開発しました。また、次世代シークエンサーの容量を大きく超える、1015個(1,000兆個)の核酸分子を計数できる可能性も示しました。さらにこの方法により、複数の試料由来の結果が混ざる問題も解決しました。
本成果により今後、がん細胞、細菌叢、ウイルスの研究やリキッドバイオプシー注4)などにおいて、より高精度な解析の実現が期待できます。
本成果は、英国の科学雑誌『Scientific Reports』に掲載されるのに先立ち、オンライン版(10月19日付け)に掲載されます。
本研究は科学技術振興機構(JST)戦略的創造研究推進事業 個人型研究(さきがけ):「統合1細胞解析のための革新的技術基盤」研究領域(研究総括:浜地 格(京都大学 大学院工学研究科 教授)における研究課題「生体システム理解・医科学応用を実現する1細胞核酸計測技術の開発」(研究者:城口 克之)の一環として行われました。
- ※1) 共同研究チーム
理化学研究所 生命システム研究センター オミックス動態研究ユニット
ユニットリーダー(JST さきがけ研究者) 城口 克之(シログチ カツユキ)
基礎科学特別研究員 小川 泰策(オガワ タイサク)
東海大学 医学部 基礎医学系 分子生命科学
教授 今西 規(イマニシ タダシ)
奨励研究員 キリル・クリュコフ(Kirill Kryukov)
- ※2) Shiroguchi, K., Jia T.Z., Sims P.A., Xie X.S. “Digital RNA sequencing minimizes sequence-dependent bias and amplification noise with optimized single molecule barcodes”Proc. Natl. Acad. Sci. USA. 109, 1347-1352 (2012).
<背景>
次世代シークエンサーは、一度にたくさんの核酸(DNAおよびRNA)の配列を決定することができます。この装置は、例えば、遺伝子発現量の指標となる細胞内のRNAの数について、多種類の遺伝子を同時に数えることもできます。近年では、1つの細胞の中に存在する多数のRNAも網羅的に計数されており、微量のRNAをより正確に計数する手法の開発が求められています。
また、多くの細胞や試料からのDNAを一度に計数するために、シークエンシング(配列決定)をする前に、試料ごとに異なるDNAタグ(6~8塩基配列程度)を付加するマルチプレックス法注5)が用いられています(図1)。しかし、配列決定の結果を得るまでの間に、試料とタグのペアが入れ替わる現象が見つかっており、正しい結果を得られないという問題がありました。
城口ユニットリーダーらは2012年に、次世代シークエンサーを利用した「DNA分子バーコード法」を開発しました。この方法は、数えたい目的の核酸分子(DNAおよびRNA)1つ1つに、それぞれ異なる配列を持つDNA分子(DNA分子バーコード、8~20塩基配列程度)を付加することで、目的の核酸分子を数えることができます(図2)。DNAバーコード法では、正確にデジタル計数することができるため、遺伝子発現量の解析などに応用され、これまでに多くの研究者や企業に利用され、かつ改良もされてきました。しかし、従来のDNA分子バーコード法では、次世代シークエンサーの配列決定でのエラーなどが原因で、100個程度の分子までしか正確に数えられないという課題や、試料とタグのペアが入れ替わることに起因する複数の試料由来の結果が混ざるという問題がありました。
<研究手法と成果>
共同研究チームは、独自のDNA分子バーコードを設計して配列決定時などのエラーを検出したり、DNAバーコードの長さをコンピュータ上で変えるなどして計数の精度を確かめることなどにより、実験と配列情報解析法を組み合わせて、従来よりも多くの分子を正確に計数できる手法を開発しました。この手法による計数の正確さを確かめるため、既知の数の合成DNAを複数準備して次世代シークエンサーを用いて計数したところ、その計数結果が合成DNAの数とほぼ一致することが示されました(図3)。
また一度の計数で、1万個以上の核酸分子を正確に数えられることが分かりました。さらに、今回開発したDNA分子バーコード法を使うと、マルチプレックス法における試料とタグのペアの入れ替わりを見つけることができ、複数の試料由来の結果が混ざる問題が解決されました。
今回達成した1万個以上の計数という成果は、従来の手法と比べて100倍以上も多い数になります。さらに、長さの異なるバーコードがそれぞれ計数できる最大分子数の値から推定することで、最大1015個の(1,000兆個)核酸分子をも数えることができる十分な可能性も示しました。この値は次世代シークエンサーの容量(107~1010個、1000万~100億個)を大きく超えるもので、より多くの分子を計数する目的において、今回開発されたDNA分子バーコード法は十分な役割を果たすことができます。
<今後の期待>
従来のDNA分子バーコードは、がん細胞、細菌叢、ウイルスの計数、リキッドバイオプシーなどへの応用研究が進んでいます。本研究で開発した分子バーコード法の応用により、より高精度の解析の実現が期待できます。
<参考図>
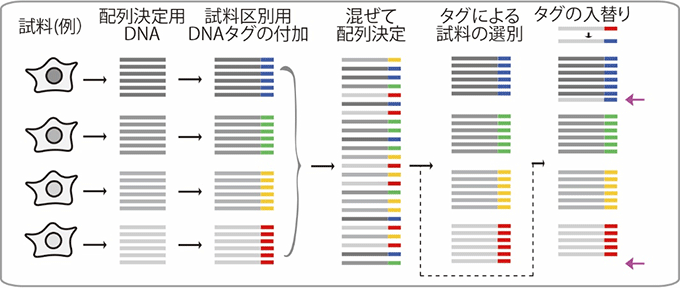
図1 複数のDNA試料を一度に配列決定するマルチプレックス法
試料から配列決定をするDNA(灰色)を準備し、試料ごとに異なるDNAタグ配列(カラー表示)を付加する。たくさんの試料由来のDNAを混ぜて一度に配列を決定し、同時に配列を決めたタグ配列により、どの試料由来のDNAであったかを識別する。近年、配列を決定する一連の過程でタグ配列が入れ替わってしまうことが報告されており(一番右の列の矢印)、問題となっている。
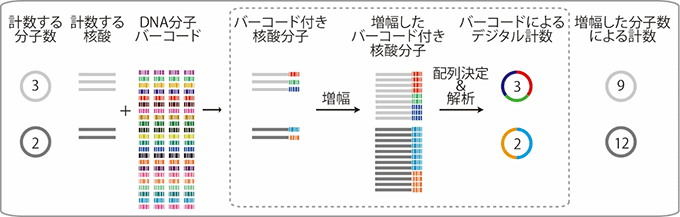
図2 DNA分子バーコード法を用いた核酸分子のデジタル計数法
配列の異なるたくさんのDNA分子バーコード(異なる色が異なる配列を示す)を準備し、数えたい核酸分子に配列の異なるDNA分子バーコードを付加する。次に、増幅した分子の配列を次世代シークエンサーで決定する。もともと存在した分子の数を決定するために、増幅された分子中のバーコードの種類の数を計数する。増幅された分子の数ではなく、各バーコード配列の有無を調べるため、デジタル計測となる。本研究では、合成されたバーコード付き核酸分子を準備し、その分子数を計数できることを示した(点線枠の部分)。
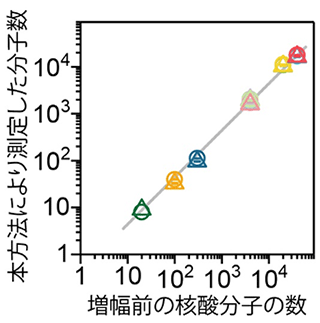
図3 増幅前のバーコード付きDNAの分子数と開発した手法で測定した分子数の関係
図2の点線枠を行った実験結果。増幅前のDNA分子数と、開発した手法により測定したDNA分子数がよく一致していることが分かる。一度に準備した複数種類のDNA分子をそれぞれ異なる量を用いて増幅し、シークエンシングにより分子数を決定した(図1の点線枠参照)。横軸の値は、増幅前のDNA分子数を、それぞれの色は異なる配列をもつDNA分子であることを、○と△の記号は2回実験を行ったことを示す。右上の黄色や赤の記号はどちらも、DNA分子数が1万個を超えていることを表している。
<用語解説>
- 注1) 次世代シークエンサー
- サンガー法を利用した蛍光キャピラリーシークエンサーである「第一世代シークエンサー」と対比させて使われる用語。機種によるが、一度に107~1010(1,000万~100億)個のDNAの配列を決定できる。以前はヒトのゲノム配列を決定するのに14年程度かかったが、次世代シークエンサーを用いると、数日で決定できる。
- 注2) DNA分子バーコード法
- それぞれ異なる配列を持つDNA分子(DNA分子バーコード)を、目的の核酸分子(DNAやRNA)の1つ1つに付加することで、核酸分子の数をデジタル的に正確に数えることができる方法(図2参照)。
- 注3) デジタル計数
- シグナルの量にかかわらず、シグナルの有無を1または0で計数する方法。今回の例では、増幅後にある分子バーコードが検出されたら、その分子バーコードを持つDNA分子が増幅前に1つ存在したとし、増幅後に分子バーコードが検出されなかったら、その分子バーコードを持つDNA分子が増幅前に存在しなかったとして計数する(図2参照)。
- 注4) リキッドバイオプシー
- 主にがんの領域で、内視鏡や針を使って腫瘍組織を採取する従来の生検(biopsy)に代えて、体液サンプル(血液など)を使って診断や治療効果予測などを行う方法。
- 注5) マルチプレックス法
- 多くの試料(細胞など)由来の配列決定用DNAを一度に配列決定するための方法。配列決定用DNAに、試料ごとに異なるDNAタグ(6~8塩基配列程度)を付加し、すべての試料由来の配列決定用DNAを混ぜて配列決定をする。配列決定度、DNAタグの配列から、核配列決定用DNAがどの試料由来であったかを識別する。
<論文情報>
| タイトル |
“The efficacy and further functional advantages of random-base molecular barcodes for absolute and digital quantification of nucleic acid molecules” |
| 著者名 |
Ogawa Taisaku, Kirill Kryukov, Tadashi Imanishi, Katsuyuki Shiroguchi |
| doi |
10.1038/s41598-017-13529-3 |
<お問い合わせ先>
<研究に関すること>
城口 克之(シログチ カツユキ)
理化学研究所 生命システム研究センター オミックス動態研究ユニット ユニットリーダー
Tel:06-6155-0834 Fax:06-6155-0112
E-mail:
<JST事業に関すること>
川口 哲(カワグチ テツ)
科学技術振興機構 戦略研究推進部
Tel:03-3512-3525 Fax:03-3222-2064
E-mail:
<報道担当>
理化学研究所 広報室 報道担当
Tel:048-467-9272 Fax:048-462-4715
E-mail:
科学技術振興機構 広報課
〒102-8666 東京都千代田区四番町5番地3
Tel:03-5214-8404 Fax:03-5214-8432
E-mail: