ポイント
- すべてのDNA解析方法において、外来DNAの混入は重大で防ぎづらい問題だった。
- 空気中を浮遊する微粒子が主な汚染源であり、実験器具の静電気も重大な要因であることを明らかにした。
- DNAの混入をほぼ完全に防止できる、DNA増幅用の卓上型クリーンルームを開発した。
JST 戦略的創造研究推進事業において、広島大学の高橋 宏和 研究員および岡村 好子 准教授らは、外来DNAの混入を防止し、信頼性の高い極微量DNA解析を可能にする卓上型クリーンルームを開発しました。
従来、DNA増幅を行う場合には、外来DNAの混入(コンタミネーション)を防止するために、主に無菌操作用のクリーンベンチが使用されていました。しかしながら、外来DNAの混入を完全に防ぐことは難しく、1細胞レベルの極微量のDNA、特に全ゲノムを増幅注1)する場合には大きな問題となっていました。
本研究グループは、従来の実験環境において、注意深く全ゲノム増幅の操作を行った場合でも生じてしまうコンタミネーションの主原因が、実験環境中の浮遊微粒子(主成分は直径0.1µm)であること、また実験器具に発生する静電気が外来DNAを含む浮遊微粒子を集めてしまうことを、明らかにしました。
さらに、主に半導体などの研究用に用いられる、国際標準規格ISO-1の清浄度(全微粒子数が10個/m3以下、図1)の空間を形成する既存のクリーンシステムをベースに、外来DNAの混入をほぼ完全に防止してDNA増幅を行うことができる新しい卓上型クリーンルームを開発しました。
開発した卓上型クリーンルームは、培養できない微生物注2)からの有用遺伝子探索など、さまざまな基礎研究分野での遺伝子解析のみならず、ガン研究などにも貢献することが期待されます。
本研究は、農研機構(国立研究開発法人 農業・食品産業技術総合研究機構)の小堀 俊郎 研究員らとの共同で、興研株式会社と関東化学株式会社の協力を得て行いました。本研究成果は、2016年7月12日(米国東部時間)に米国科学誌「BioTechniques」のオンライン速報版で公開されます。
本成果は、以下の事業・研究領域・研究課題によって得られました。
戦略的創造研究推進事業 チーム型研究(CREST)
| 研究領域 |
「藻類・水圏微生物の機能解明と制御によるバイオエネルギー創成のための基盤技術の創出」
(研究総括:松永 是 東京農工大学 学長) |
| 研究課題 |
「海洋微生物発酵制御を基盤とした大型藻類の完全資源化基盤技術の開発」 |
| 研究代表者 |
中島田 豊(広島大学 教授) |
| 研究期間 |
平成24年10月~平成30年3月 |
JSTはこの研究領域で、高い脂質・糖類蓄積能力や多様な炭化水素の産生能力などを持つものがある藻類・水圏微生物に着目し、これらを活用したバイオエネルギー創成のための革新的な基盤技術の創出を目指しています。上記研究課題では、海洋微生物が持つ耐塩性および海藻糖質代謝機能に着目し、海洋藻類のエネルギー・資源化システム実用化に必要な要素技術の確立を目指しています。
<研究の背景と経緯>
DNAの増幅と解析は、すでに基礎研究のみならず犯罪捜査に至るまでさまざまな分野で利用されています。しかし、目的試料への外来DNAの混入(コンタミネーション)は、DNA解析において重大かつ防ぎづらい問題として認識されながらも、原因の特定が困難で解決が難しい問題でした。微量のDNAを増幅するPCR法は高精度・高感度であるにも関わらず、臨床診断ではコンタミネーションによる誤診を防ぐために、結果を判断する際に他の検査方法と併せる必要があります。また、次世代シークエンサー注3)による核酸配列の大規模解析でも、試料とは無関係なDNA配列の混入が起きていることが報告されており、データの信頼性の問題が指摘されています。
これまで、作業者の経験・技量不足や、遺伝子組換えで作製された酵素へのDNAの混入注4)がコンタミネーションの主原因であると考えられていました。一方で、増幅試薬の調製段階における環境、特に空気中に浮遊している微粒子が汚染原因になり得ることがあることも報告されています。そのため無菌操作に使用するクリーンベンチ(国際標準規格ではISO-5、図1)が、一般的にはDNA増幅用の試薬調製に利用されています。しかし、微生物やヒトの1細胞から全ゲノム増幅を行う場合、クリーンベンチを使用してもコンタミネーションは防止できず、DNAを加えなかった試料からもDNA増幅が生じてしまうため、結果の再現性および信頼性が著しく低下してしまいます。
本研究チームでは、培養できない微生物の有用遺伝子の利用を想定しており、このような1細胞レベルのDNA増幅が必要になる先端的研究を行う場合には、コンタミネーションの問題を解決して、信頼性の高いデータを得ることが必要です。
そこで、DNAコンタミネーションの原因を特定し、それを防止する方法の開発を進めました。
<研究の内容>
一般的なクリーンベンチ(ISO-5環境)では、主に粒径0.1µmの微粒子が2.0〜3.5万個/m3も存在しています。この粒径はウイルスの大きさに相当し、事前の報告からこの微粒子のうちおよそ100〜1000個がウイルス様粒子と推定されており、コンタミネーションの主な原因と予想していました。しかし、クリーンベンチで調製した試料に混入したDNAから増幅されたDNAの配列を決定すると、予想に反してヒト配列の検出頻度が高いほか、微生物由来のDNAも確認されました。また、コンタミネーションを起こす頻度や汚染源にはまったく規則性がなく、実験場所(広島大学、農研機構、関東化学)によってもコンタミネーションを起こす頻度、ヒト由来以外のDNA配列の種類は異なっていました。0.5µm以上の浮遊微粒子がほとんど存在していないISO-5環境では、おおよそ10µmの大きさであるヒト細胞は空気中を浮遊しているとは考えにくく、死細胞から遊離した浮遊DNAを含む0.1µm粒子がコンタミネーションの主原因であると推測されました。この推測を証明するためには、粒子経0.1µmの微粒子がほとんど存在しない実験環境でDNA増幅を行う必要があります。そこで、ISO-1(図1、全微粒子数が10個/m3以下)を形成できる興研株式会社のオープンクリーンシステムを利用して、全ゲノム増幅用の卓上型クリーンルームを開発することにしました。
開発のベースとしたシステムは、整流された気流によって浮遊微粒子をクリーンエリア外に押し出して排出すると言うコンセプト(図2(1))の装置です。この装置は、半導体やナノテクノロジー研究用に開発されたもので、空気清浄度が低レベルのクリーンルーム(ISO-6〜7)内に設置することが前提になっているために、周囲が囲われていません(図2(2))。そのため、一般的な実験室に設置した場合には、システムが作動していない時は実験器具への落下菌などの付着の恐れがありました。また、作動中でも、落下してくるカビ胞子や花粉などの粒径30µm以上の重量のある飛来物は排除できない恐れもあったため、システム内の清浄エリアを覆うフードを設置しました(図3)。フードの設置自体も作業者の腕の動きも清浄度には影響せず、ISO-1環境を維持可能なことが確認できました。そこで、このフードを設置した卓上型クリーンルームシステム(ISO-1環境)で試薬調製した場合と、一般的なクリーンベンチ(ISO-5環境)で調製した場合で、同一の試薬を用いた全ゲノム増幅へのコンタミネーションを比べたところ、ISO-5でのみコンタミネーションが確認され(図4)、粒子経0.5µm未満の浮遊微粒子中にDNAが含まれていることが明らかとなりました。
一方で、卓上型クリーンルームを用いて全ゲノム増幅実験を繰り返し行うと、実験回数の増加とともに、クリーンルーム内はISO-1を維持しているにもかかわらず、コンタミネーションの頻度が上昇してきました。汚染源についてさまざまな検討を行ったところ、実験中の気流との摩擦によってプラスチック製実験器具の表面に静電気が発生し、実験が終了してシステムを停止したときに流入してくる空気に含まれる微粒子が、その静電気によって付着することが確認されました(図5)。さらに、一度付着した微粒子は除電されてもプラスチック表面に付着したままとなることも確認されました。そこで装着したフード内面の天井に除電器を設置し(図3)、実験中に発生する静電気を速やかに除去したところ、コンタミネーションをほぼ完全に抑制することに成功しました(図6)。実験器具・道具に発生した静電気が、微量の試薬や試料を飛び跳ねさせるなどの問題は報告がありましたが、DNAコンタミネーションへの影響については、世界で初めての報告です。
<今後の展開>
今回開発した卓上型クリーンルームシステムは、興研株式会社から国内向けに販売が開始されています。本クリーンルームによって、1細胞由来の極微量のDNAから高い信頼性と再現性を持った全ゲノム増幅が可能になるため、これまで困難であった培養できない微生物の有用遺伝子探索研究のさらなる促進が期待できます。また、全ゲノム増幅法はヒトの1細胞からでも可能なため、末梢血中の遊離ガン細胞(Circulating tumor cell、CTC)や血中遊離DNA(cell-free DNA、cfDNA)の解析注5)などの医学研究への応用や、臨床におけるDNA解析への応用など、幅広い分野での利用が期待できます。
<参考図>
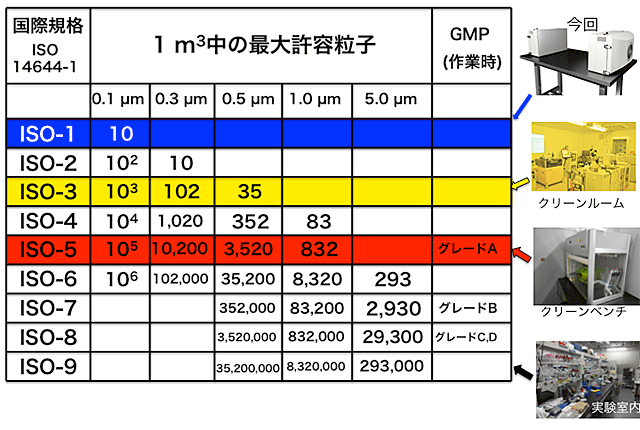
図1 クリーンルームの国際規格
空気清浄度は空気1m3中の0.1µm以上の浮遊粒子数で規定される。一般的な実験室はISO-9に分類され、ライフ・バイオ分野で汎用されているクリーンベンチはISO-5であり、浮遊微粒子の主成分は直径0.1-0.3µmの粒子で、0.5µm以上の微粒子はほとんど検出されない。

図2 卓上型オープンクリーンシステムの原理
(1) クリーンゾーンの両側に設置するプッシュフードには超高性能フィルターが組み込まれている。このフィルターを通過し清浄化された、風速と風向がすべて同様に調整されている気流(矢印)が、2つのプッシュフードから互いに向かって吹き出される。この気流がフード間の中央でぶつかり合い、常にクリーンゾーン(青色)の外側へ向かう気流が形成される。
(2) その結果、2つのプッシュフードに挟まれた空間は高い清浄度を保ち続けるため、周囲を囲うことなくクリーンゾーンを形成することが可能である。

図3 DNA増幅用ISO-1クリーンルーム
2つのプッシュフード(PH1、2)に挟まれ、飛来物防止フードに囲まれた空間の清浄度はISO-1(0.1µm以上の微粒子が10個/m3以下)のクリーンエリアになる。使用時は除電器で除電しつつ、パーティクルカウンターで微粒子数を計測しながら、DNA増幅用反応液の調製を行う。実験器具などは、汚染を避けるためにクリーンエリア内で保管する。
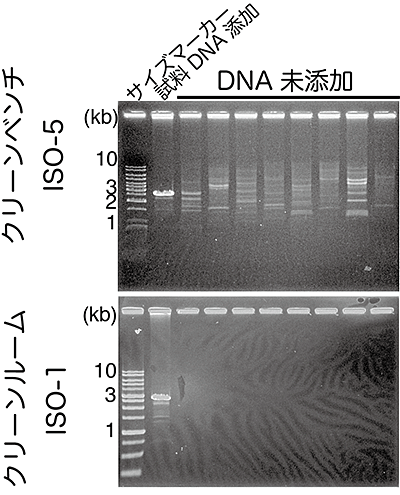
図4 クリーンルーム使用による浮遊微粒子汚染の防止
同一の試薬を用いたDNA増幅の結果。白い線はDNAを示す。クリーンベンチ(ISO-5)で行った実験(上図)では試料DNA未添加の場合でも、増幅DNA由来の縞模様が現れており、コンタミネーションを起こしてしまったことが分かる。一方、今回開発した卓上型クリーンルーム(ISO-1)を使用した場合(下図)は、増幅DNA由来の縞模様が現れておらず、混入が防止できている。

図5 静電気の発生
システムが稼働中は、気流との摩擦によって、プラスチック製品の表面上に静電気が発生(摩擦帯電)する。システムが停止すると外部から流入した微粒子が排出されないため、帯電したプラスチック製品には電気集塵作用により微粒子が付着してしまう。この付着微粒子が、以降の実験へのDNA汚染源となり得る。この結果を踏まえて、清浄空間内部に、除電器(図3、黄矢印)を設置し、システム稼働中に起きる、プラスチック製品の帯電を防止した。
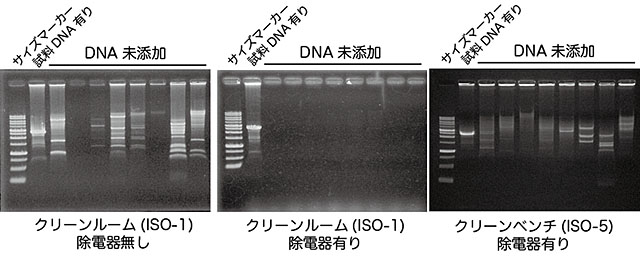
図6 除電器による除電の効果
同一の試薬を用いて、除電器の有り無しで繰り返しDNA増幅実験を行った結果の一部。除電器を設置していない卓上型クリーンルームで行った実験(左図)では、ISO-1環境であるにもかかわらず、試料DNA未添加の場合でも増幅DNA由来の縞模様が現れており、静電気によって付着した微粒子がコンタミネーションを起こすことが分かる。一方、清浄空間内部に除電器(図3、黄矢印)を設置した卓上型クリーンルームを使用した場合(中央図)は、増幅DNA由来の縞模様が現れておらず、安定して混入が防止できている。また、ISO-5のクリーンベンチに除電器を設置した場合(右図)でも、増幅DNA由来の縞模様が現れており、環境をISO-1に保つ必要性が再確認された。
<用語解説>
- 注1) 全ゲノム増幅法
- 特殊なDNA合成酵素を用いてゲノムDNAを大量に増幅する方法で、微生物やヒトの1細胞からでも、全ゲノム配列の解析が可能になると期待されている。しかし、非特異的にDNAを増やすため、特異的に増やすPCRよりも混入したDNAが増幅しやすいことが分かっている。
- 注2) 培養できない微生物
- 地球上の全微生物の99%以上は培養困難であると言われている。この難培養微生物の遺伝子を直接探索できれば、より多くの有用物質を発見できる可能性があるが、1細胞由来の極微量のゲノム全体を大量に増幅する必要がある。
- 注3) 次世代シークエンサー
- これまで使用されていたDNA配列決定装置(シークエンサー)と比較して、安価に高速で大量の配列解析が可能な装置の総称。ヒトゲノムでも1ヵ月以内で解析が可能なため、現在医療現場での活用が進められており、精密医療(患者の腫瘍に発生した正確な突然変異の特定など)の分野で期待が高い。
- 注4) 酵素にDNAが混入
- 一般的に分子生物学で使用される酵素は、組換え大腸菌で作成される。そのためDNAに親和性が高い酵素、例えばPCRに使用する耐熱性ポリメラーゼには大腸菌ゲノムが混入している。本研究では、これまでに開発したDNAの混入を極限まで減らした特別なDNA-free酵素(関東化学から販売中)を使用している。
- 注5) CTCおよびcfDNA解析
- CTCは転移可能な腫瘍組織から血中に浸潤してきた細胞と定義されている。cfDNAはcirculating tumor DNA(ctDNA)とも呼ばれ、免疫反応などによるガン細胞の死滅によって血中に流れ出てくるDNAと定義されている。両解析ともに、ガンの病態予測に重要であると考えられており、現在研究が進められている。
<論文情報>
“Development of a bench-top extra-cleanroom for DNA amplification”
(DNA増幅のための卓上型エクストラクリーンルームの開発)
doi :10.2144/000114433
<お問い合わせ先>
<研究に関すること>
岡村 好子(オカムラ ヨシコ)
広島大学 大学院先端物質科学研究科 准教授
〒739-8530 広島県東広島市鏡山1−3−1
Tel:082-424-4583 Fax:082-424-4583
E-mail:
<JST事業に関すること>
川口 哲(カワグチ テツ)
科学技術振興機構 戦略研究推進部
〒102-0076 東京都千代田区五番町7 K’s五番町
Tel:03-3512-3524 Fax:03-3222-2064
E-mail:
<報道担当>
科学技術振興機構 広報課
〒102-8666 東京都千代田区四番町5番地3
Tel:03-5214-8404 Fax:03-5214-8432
E-mail:
広島大学 社会産学連携室 広報部 広報グループ
〒739-8511 広島県東広島市鏡山1-3-2
Tel:082-424-6781 Fax:082-424-6040
E-mail:
農研機構 食品研究部門 企画管理部 交流チーム
〒305-8642 茨城県つくば市観音台2-1-12
Tel:029-838-7980 Fax:029-838-8005
E-mail:
興研株式会社 環境 エンジニアリングディビジョン
〒102-8459 東京都千代田区四番町7番地
Tel:03-5276-1931 Fax:03-3265-1976