国立遺伝学研究所 分子機能研究室の鐘巻 将人 准教授らのグループは、「ヒト培養細胞」で特定のタンパク質を素早く分解除去する方法を開発しました。これまでモデル生物注1)でしかできなかった精緻な遺伝学研究が、ヒト細胞でもできるようになる画期的な手法です。
利用したのは、同グループが2009年に開発した「AID法注2)」というタンパク質分解除去システムです。これは、動物細胞の特定のタンパク質を分解除去し、そのタンパク質を司る遺伝子の役割を解析できるシステムです。このシステムをヒト細胞にも導入できるよう、ゲノム編集注3)技術を応用し、ヒト細胞内のタンパク質の分解除去を自在に操ることに成功しました。
今回開発した手法を使うと、これまで数日を要していたタンパク質の分解除去が、1時間程度で行えるようになり、細胞分裂や増殖などの、細胞の基本現象に与える影響を直接観察することが可能になります。また新技術では、分解除去のタイミングも任意に設定できるため、今までの技術では解明が難しかった、生存に不可欠な「必須遺伝子」の機能解析ができるようになります。本技術により今後がん細胞を含めた、ヒト細胞が増殖するための仕組みの解明が大いに進むことが期待されます。
本研究成果は、2016年3月24日(米国東部時間)に米国科学誌Cell Reportsオンライン版に掲載されます。
<研究の背景>
私たちの体を形作る基本最小単位である細胞が、どのように生命活動を維持し、増殖するかを理解するためには、遺伝子の情報に基づいて作られる個々のタンパク質の機能を理解することが欠かせません。細胞内におけるタンパク質の機能を理解するためには、特定のタンパク質が消失した際の影響を観察することがとても有効です。これまでの研究では、短鎖干渉RNA(siRNA注4))を利用したタンパク質発現抑制がよく使われてきましたが、タンパク質の消失に数日もの時間を要します。また、近年開発されたCRISPR/Casを利用した遺伝子ノックアウト(遺伝子破壊)細胞も研究に使われるようになってきましたが、細胞の中には10%程度の“ノックアウトできない遺伝子(必須遺伝子)”が存在します。これらの状況から、現在の技術では機能の解明が難しいタンパク質が数多く存在しています。
<本研究の成果>
本グループは植物細胞が持つオーキシン依存的タンパク質分解の仕組みをヒト培養細胞に移して、任意のタンパク質をオーキシン添加時に消失させるオーキシンデグロン注5)(AID)法を2009年に開発しました。この方法の特徴は、目的とするタンパク質を1時間程度で消失させるだけでなく、スイッチのように可逆的にタンパク質の「ある」「なし」を制御することが可能であることです。一方で、AID法を行うためには、標的タンパク質の情報を持つ遺伝子を人為的に改変し、目印となる「分解タグ」の情報を持つDNA配列を導入する必要があります。そこでCRISPR/Casゲノム編集技術を応用して、分解タグを簡便に導入する技術を開発することに成功しました。これらの技術を組み合わせることにより、任意のタンパク質の「ある」「なし」を自在に制御できる技術、すなわち「AID変異細胞の作成法」を完成させました。また、この度開発したタグ導入技術を利用して、蛍光タンパクタグなども任意の遺伝子に導入することが可能になりました。
<今後の期待>
今回開発した技術は、特にヒト細胞が生きていくために必須な“ノックアウトできない”遺伝子(全遺伝子質のうち10%程度存在する)から作り出される重要なタンパク質の機能解明に威力を発揮します。さらに、タンパク質の除去が迅速であるという特徴は、タンパク質消失が細胞分裂や増殖などの短時間で状態が移り変わる、細胞の基本現象に与える影響を観察することを可能にします。今後本技術を世界中の研究者が利用することにより、がん細胞を含めたヒト細胞における様々な仕組みが解明されることが期待されます。また、本技術はヒト培養細胞以外の様々な細胞にも応用できる可能性があります。実際に私たちはマウスES細胞への技術応用を進めており、今後はさらに様々な生物種の細胞でも広く利用可能になることを期待しています。
<研究体制と支援>
本研究は、清光 智美 助教(名古屋大学)、相賀 裕美子 教授(遺伝研)との共同研究成果です。本研究の遂行にあたり、科学技術振興機構(JST) 戦略的創造研究推進事業 個人型研究(さきがけ)(鐘巻、清光)、遺伝研共同利用研究費(清光、2014-B、2015-A1)および科研費による支援を受けました。本研究で作成した研究材料は全てナショナルバイオリソースから配布し、世界中の研究者が利用できます。
<参考図>
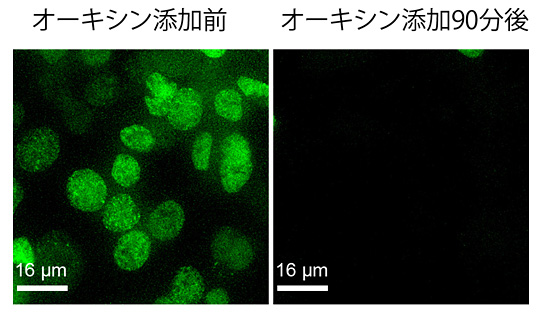
図1
オーキシン添加前後の核に局在するコヒーシンタンパク質の発現を蛍光法により観察した結果、添加90分後の細胞では、コヒーシンタンパク質が消失した。
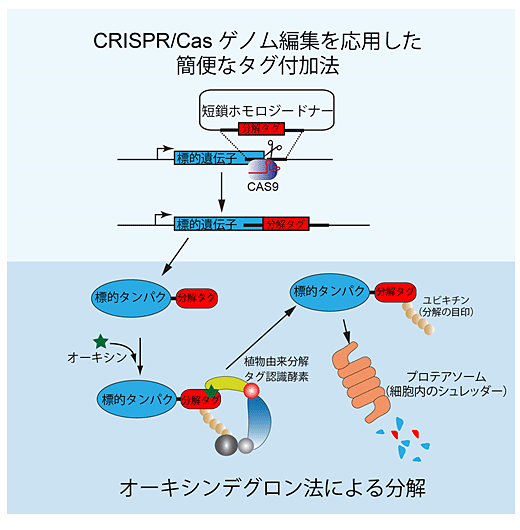
図2
CRISPR/Casゲノム編集を利用したタグ付加とオーキシンデグロンの作用機序を示したモデル図。上段に新たに開発したCRISPR/Casを利用した目印となる分解タグの導入法、下段にAID法の作用機序を示している。
<用語解説>
- 注1) モデル生物
- これまで細胞分裂や増殖などの基本的な仕組みは、ヒト細胞から酵母までほぼ共通であるという認識のもとで、より研究材料として扱いやすい“モデル”となる生物が基礎研究に使われてきた。しかし、生命医学的視点からはヒト細胞における研究が欠かすことができなかった。
- 注2) AID法
- Auxin-Inducible Degron(オーキシンデグロン)法の略。植物由来のオーキシン依存のデグロン配列を分解の目印として任意のタンパク質に付加することにより、オーキシン添加時にそのタンパク質を消失させることを可能とするオリジナル技術。
- 注3) ゲノム編集
- CRISPR/Casに代表されるゲノム編集は任意のDNA配列を切断することを可能にする。これにより特定の遺伝子座の改変が可能になった。その可能性から大いに注目されている技術。
- 注4) siRNA
- 短鎖干渉RNA。目的のタンパク質の情報を持つmRNAに対して、相補的結合する短鎖RNAを合成し、これを細胞に導入することで細胞内のRNA干渉機構を利用してタンパク質発現を抑制する技術。
- 注5) デグロン
- 分解を引き起こす目印となるアミノ酸配列。
<論文タイトル>
“Rapid Protein Depletion in Human Cells by Auxin-Inducible Degron Tagging with Short Homology Donors”
(短鎖ホモロジー領域を持つドナーDNAによるタグ付加を利用したヒトオーキシンデグロン変異細胞の構築)
著者:Toyoaki Natsume, Tomomi Kiyomitsu, Yumiko Saga, and Masato T. Kanemaki(夏目 豊彰、清光 智美、相賀 裕美子、鐘巻 将人)
doi :10.1016/j.celrep.2016.03.001
<お問い合わせ先>
<研究に関すること>
鐘巻 将人(カネマキ マサト)
情報・システム研究機構 国立遺伝学研究所 新分野創造センター 分子機能研究室 准教授
Tel:055-981-5830
E-mail:
URL:https://www.nig.ac.jp/labs/MolFunc/Jpn/
<JST事業に関すること>
川口 哲(カワグチ テツ)
科学技術振興機構 戦略研究推進部 ライフイノベーショングループ
Tel:03-3512-3525
E-mail:
<報道担当>
情報・システム研究機構 国立遺伝学研究所 リサーチ・アドミニストレーター室
Tel:055-981-6745
E-mail:
科学技術振興機構 広報課
Tel:03-5214-8404 Fax:03-5214-8432
E-mail: