ポイント
- 遺伝子発現量を蛍光シグナルで検出する高速化学反応を開発
- 酵素を使わず、一定温度で遺伝子シグナルを1,500倍に増幅し高感度検出
- 次世代の技術として期待される、生細胞内遺伝子発現解析法への応用が可能
JST 課題達成型基礎研究の一環として、北海道大学 大学院薬学研究院の阿部 洋 准教授(前 理化学研究所 伊藤ナノ医工学研究室 専任研究員)らは、遺伝子発現の情報を生きた細胞内で化学的に増幅して検出する分子プローブを開発しました。
細菌検査や医療診断などで、遺伝子解析技術の利用が進んでいます。現在、遺伝子の発現量を調べる方法として一般に用いられているリアルタイムPCR法注1)は、1)細胞を破壊してRNAを抽出する、2)RNAからDNAに変換する、3)温度サイクルによりDNAを増幅する――それぞれの過程で時間とコストがかかります。このため、安価で簡便迅速な遺伝子検出技術の開発が期待されています。
今回、阿部准教授らは、細胞に含まれる特定の遺伝子の発現の有無を、酵素を用いず一定温度で、生きた細胞内で化学増幅して検出できるプローブの開発に成功しました。このプローブは、従来の方法とは異なり、細胞と混ぜるだけで遺伝子の発現の有無を解析でき、解析にかかるコスト、時間、スペースを節約できることから、環境細菌検査や医療診断など、その場で検査が必要な技術への応用が期待されます。
本研究は、岐阜大学 工学部 生命工学科の柴田 綾 特任助教(前 理化学研究所 伊藤ナノ医工学研究室 基礎科学特別研究員)、理化学研究所 伊藤ナノ医工学研究室の鵜澤 尊規 研究員、伊藤 嘉浩 主任研究員、北海道大学 大学院薬学研究院の周東 智 教授と共同で行ったものです。
本研究成果は、米国化学会誌「Journal of the American Chemical Society」のオンライン速報版で近日中に公開されます。
本成果は、以下の事業・研究領域・研究課題によって得られました。
戦略的創造研究推進事業 個人型研究(さきがけ)
| 研究領域 |
「脳神経回路の形成・動作と制御」
(研究総括:村上 富士夫 大阪大学 大学院生命機能研究科 特任教授) |
| 研究課題名 |
「シナプス可塑性に関わるRNA群の革新的イメージング法の開発」 |
| 研究者 |
阿部 洋(北海道大学 大学院薬学研究院 准教授) |
| 研究期間 |
平成23年10月~平成27年3月 |
JSTはこの領域で、脳の統合的理解を目指し、新たな視点に立って脳を構成する神経回路の形成やその動作原理並びにその制御機構の解明に挑戦する研究を対象とします。上記研究課題では、脳が働くための基盤となる脳神経回路がどのようにして形成されるかという問題に対して、細胞内機能ドメインの役割から解析し、脳の形成機構およびその破綻による脳疾患の機構に迫ります。
<研究の背景>
細菌検査や医療診断などで、遺伝子解析技術の利用が進んでいます。リアルタイムPCR法は、DNAを温度サイクル装置(PCR装置)で増幅し、蛍光プローブを用いて高感度に検出する技術です。細胞からmRNAを抽出し、逆転写して得られたDNAをリアルタイムPCRで検出すれば、細胞中の遺伝子の発現量を解析することができます。しかし、リアルタイムPCRの問題点として、RNAを細胞から取り出してDNAに変換するための時間とコストがかかること、細胞からRNAを抽出する際にRNAが壊れるなどして情報が失われる恐れがあることなどが挙げられます。そのため、細胞からRNAを抽出するのではなく、直接生きた細胞内で、特定のRNAを検出する方法が開発できれば、次世代の画期的な遺伝子診断法になることが期待されます。しかしながら、細胞中に数コピーしかないRNAの情報を、細胞が死なない温和な条件下で蛍光シグナルとして増幅する、これまでにない技術が必要になります。
<研究の内容>
今回、阿部准教授らは、生細胞中で、酵素を用いず標的RNAを認識して蛍光シグナルを発するプローブを設計、開発し、実際に生細胞内で蛍光シグナルが検出できることを実証しました。
(1)プローブのシグナル発生原理
生細胞中に数コピーしかないRNAを検出するには、検出シグナルを大幅に増幅する必要があります。このため、開発する蛍光プローブには、RNAと結合した後速やかに蛍光を発する、蛍光を発した後は速やかにRNAから離れて、次のプローブが反応する余地を作るという特徴が必要です。そうすれば、1分子のRNAに対して多数のプローブが反応するので大きな蛍光シグナルを得ることができます。しかし、従来型の検出プローブは化学反応速度が非常に遅いため、十分にシグナルを増幅することができませんでした。阿部准教授らはこの問題の解決を試み、非常に化学反応速度の速い遺伝子検出法として芳香族求核置換反応注2)を利用した系を開発しました。このプローブは2-シアノ-4-ニトロベンゼンスルホニル(CNs)基で保護したアミノクマリン(AMCA)注3)を修飾したCNs-AMCAプローブと、チオフェノール基で修飾されたMBAプローブの2本1組からなります。プローブの端には任意の配列のDNA鎖(あるいはRNA鎖)を結合させることができます。DNAやRNAを構成する4つの塩基はそれぞれアデニンとチミジン(ウラシル)、グアニンとシトシンが必ず対になる性質があります。この性質を利用して、標的RNAに相補的なDNA鎖をプローブに連結させれば、標的RNAに2本のプローブが結合することができます。そして標的RNAに結合することでプローブ間の距離が近接すると、芳香族求核置換反応が起こってCNs基がAMCAから外れます。この反応の結果、AMCAは蛍光を発します(図1)。
(2)試験管内での遺伝子検出
次に、意図した通りに化学反応が起こってAMCAが生成されるか検討するため、特定の配列のDNA(標的DNA)と、そのDNA配列と相補的なDNA鎖を持つプローブを、濃度が1対1になるように液中で混合し、蛍光シグナルを測定しました(図2)。標的DNAが存在する条件では、時間経過とともに、化学反応が進行してAMCAが生成していました。AMCA生成率は60秒後には頭打ちになっており、反応がほぼ完結しました。一方で、標的DNAがない場合(赤線)、AMCAは生成されませんでした。以上のことから、我々が開発したプローブは従来法と比較して非常に反応速度が速いのに加え、標的が存在しない場合に副反応が起こりにくいため、シグナル/ノイズ比が高いことが分かりました。
また、低濃度の標的DNAを検出できるかを検討するため、プローブ濃度は変えずにDNAの量を変えて混合し、15時間後の蛍光を検出しました(図3)。結果、試験管内で0.5pM(生細胞内で1分子のDNAに相当)の微量の遺伝子を検出することに成功しました。また、この条件下では、15時間でDNA1分子に対して1,500個のプローブが結合して蛍光シグナルを発することが分かりました。
(3)生きた大腸菌内での遺伝子検出
このプローブを用いて、生きた大腸菌内のRNAを検出できるか試しました。標的には、細胞中に最も多く含まれるRNA配列を選びました。大腸菌の細胞壁はそのままではプローブなどの大きな分子を通さないので、大腸菌が死なない程度の薄い界面活性剤で大腸菌を処理してから、1uMプローブと10分間混合しました。その後、大腸菌の蛍光画像を顕微鏡(倍率:x60、露光時間:1秒)で撮影しました。標的RNA配列と相補的なDNA鎖を持ったプローブを混合した時に限り、AMCA由来の青色の蛍光が観察されました(図4)。一方で、ランダムな配列のDNA鎖を持ったプローブや、一方のプローブのみを大腸菌内に導入した場合には、蛍光は全く観察されませんでした。この結果から、プローブは生きた細胞内で配列選択的に遺伝子を検出できることが示されました。
界面活性剤で処理し、プローブを投与した大腸菌は、その後、培養用培地に移すことで問題なく生育することも確認しました。
<今後の展開>
今回の実験より、芳香族求核置換反応を利用したプローブが、化学増幅反応を起こすことで、標的RNAを高感度に検出できることが明らかとなりました。RNAの抽出操作やPCRによる増幅操作を必要としない次世代の細胞内遺伝子発現検出法として、あるいは生細胞内RNAイメージング技術としての応用が期待されます。さらに、実用性を高めるためには、青色のみではなく今後はさまざまな色のプローブにより同時に複数標的を観察できることが望まれます。また、本技術を用いることで、脳神経細胞内でRNAがどのように局在化しているかなどをリアルタイムで検出することが可能になります。近年、記憶が形成されるための分子基盤にRNAの動態が関わっていると考えられています。本技術により神経細胞におけるRNAの動態を解析することで得られる情報は「記憶・学習」機能がどのように起こるのかを解明するのに貢献できると考えられます。さらに、細胞内の遺伝子の発現状態を生きたまま読み取ることができれば、その細胞の2次利用も可能となります。例えば、再生医療の分野において、幹細胞から分化細胞を得る際、遺伝情報に基づいて特定の組織に分化した細胞のみを回収することによって品質を管理することが可能となるかもしれません。
<参考図>
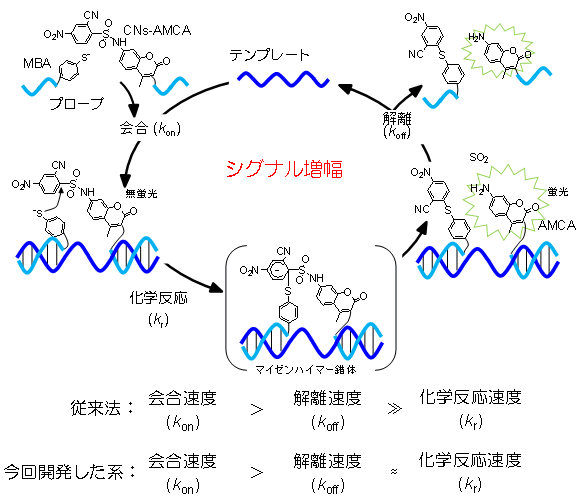
図1 鋳型反応によるシグナル増幅のイメージ図
無蛍光のプローブが1本鎖の標的核酸鎖の相補部分に結合すると、プローブ間で化学反応が起こり蛍光を発生します。その後、プローブは標的核酸鎖から解離します。反応前後でプローブと標的配列との結合力は変化しません。プローブの量を標的核酸に対し十分多く与えれば、フリーになった標的核酸鎖には別のプローブが結合します。このサイクルを繰り返すことで、蛍光シグナルを蓄積できます。
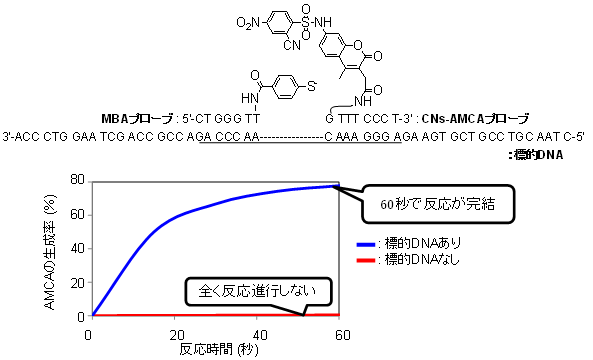
図2 芳香族求核置換反応を利用した遺伝子検出
- (上図)実験に使用したプローブと標的DNAの配列。
- (下図)AMCAの生成率の時間変化。50nMプローブと50nM標的DNAを試験管内で混合し、プローブから発生する蛍光シグナルをもとにプローブの反応収率を計算しました。結果、標的DNAが存在する場合(青線)、時間経過とともに反応が進行し、60秒後には反応がほぼ完了しました。一方で、標的DNAが存在しない場合、反応は全く進行しませんでした。
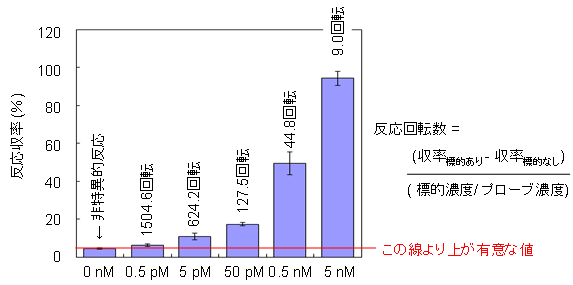
図3 微量遺伝子の検出および反応回転数
プローブ濃度を50nMに固定して、標的DNA量を変化させた場合のそれぞれのプローブの反応収率を計算しました。グラフは15時間後のプローブの反応収率と、それを基に標的DNAに対してプローブが何回反応したかを数値(反応回転数)で示しています。標的DNAが0nMの条件で得られた値が非特異的に起こった反応の収率になります。この値を閾値として反応回転数を計算しました。標的DNA量が少なくプローブが過剰となる条件下では、より多数のプローブが標的DNA上で反応することになります。この結果から、0.5pM標的DNAにおいて約1,500回の化学増幅反応が起こることで、微量な標的DNAでも有意なシグナルを検出できることが明らかとなりました。
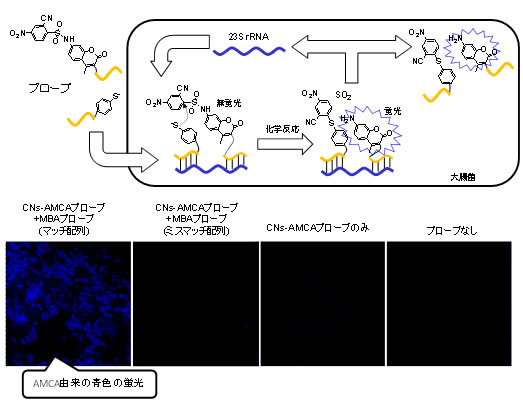
図4 大腸菌内の遺伝子検出
- (上図)大腸菌に投与した際のプローブの挙動を模式化したもの。
- (下図)生きた大腸菌に、23SrRNAを標的とするプローブを投与した。標的配列に相補なマッチ配列のプローブを用いた場合(最左)には蛍光シグナルが観察されました。一方で、標的RNAの配列に結合しないミスマッチ配列プローブを用いた場合(最左から2つ目)には蛍光シグナルは観察されませんでした。
<用語解説>
- 注1) リアルタイムPCR法
- PCR増幅産物の増加をリアルタイムでモニタリングし、解析する技術。
- 注2) 芳香族求核置換反応
- 求核置換反応の一種。電子の豊富な反応基(求核基)が芳香環の電子不足(求電子的な)部位を攻撃すること。
- 注3) アミノクマリン(AMCA)
- 蛍光化合物の一種。紫色(375nm)付近の光を当てると青色(450nm)付近の蛍光を発生する。
<論文タイトル>
“Very rapid DNA templated reaction for efficient signal amplification and its steady-state kinetic analysis of the turnover cycle”
(高速DNA鋳型化学反応によるシグナル増幅とその反応の速度論的解析)
doi: 10.1021/ja404743m
<お問い合わせ先>
<研究に関すること>
阿部 洋(アベ ヒロシ)
北海道大学 大学院薬学研究院
〒060-0812 北海道札幌市北区北12条西6丁目
Tel:011-706-3976 Fax:011-706-3769
E-mail:
<JSTの事業に関すること>
木村 文治(キムラ フミハル)、川口 貴史(カワグチ タカフミ)、眞後 俊幸(シンゴ トシユキ)
科学技術振興機構 戦略研究推進部 ライフイノベーション・グループ
〒102-0076 東京都千代田区五番町7 K’s五番町
Tel:03-3512-3525 Fax:03-3222-2063
E-mail: