|
|
九州大学大学院 総合理工学研究院 物質科学部門 青木百合子 教授
|
| |
機能性高分子や蛋白質・核酸等の生体高分子の機能・作用の本質を計算科学で解明する方法は、
分子力学法・分子動力学法(MD)等のシミュレーションを用いる方法が一般的です。
しかし、これら古典論に基づく方法では、高分子の巨視的な性質や動きを見るためには手軽に計算でき有効ですが、
原子・分子レベルで機能・作用の本質を解明するためには残念ながら限界があります。
一方で、物質を構成している個々の分子の性質をミクロな立場から明らかにする方法として、量子化学の分野で近年急速に発展を遂げた分子軌道法がありますが、
これは量子力学的に厳密に解く方法に基づいているため、分子を構成する原子数の3〜4乗に比例するという膨大な計算時間がかかってしまいます。
これでは実際のところ巨大分子の計算は時間的に不可能なので、一般的に巨大分子を簡略化し、原子数を減らした小さな分子と見なして計算する方法が取られます。
しかしながら、この方法では、計算精度を大きく犠牲にしてしまい有意な結果は得られません。
このように、小さな分子に対して成功を遂げた量子化学計算は生体高分子などの巨大分子系に対しては未だに無力なのです。
そのため、計算時間と計算精度の両方に対して満足のいく新たな方法の確立が待ち望まれています。
戦略的構造研究推進事業のさきがけ「シミュレーション技術の革新と実用化基盤の構築」領域における青木教授のプロジェクトでは、
この問題を解決するために、タンパク質あるいはDNAのような生体高分子を含む巨大高分子系に対して、原子数の1次に比例した計算時間で効率よく、
しかも精度よく、計算できる手法−を開発しました。また、Elongation法に半経験的、非経験的分子軌道計算、および密度汎関数法等に組み込むことで、
その効率性と信頼度の向上を実証できました。
|
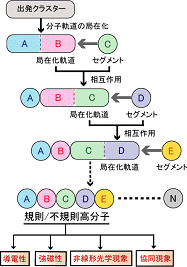
Elongation法の原理は、まず目的とする高分子鎖の一部を取り出し、一旦オリゴマー分子を作成し、
必要ならその末端を水素原子でキャップしておきます。その小さなオリゴマーを出発クラスターとして、高分子の重合反応を追跡できるようにしてセグメント
(分子ユニット)を順次追加させながら、高分子鎖の電子状態を求めていく方法です。高分子鎖にセグメントを付加させるたびに、
高分子鎖側の反応末端と付加するセグメント間の局所的な相互作用のみを取り扱うので、全系をまともに扱う従来の計算方法と比較して、
極めて効率よく電子状態を求めることができるようになりました。
|
|
例えば、ポリグリシン巨大系の演算において、従来法との演算時間を100%とすると、
本方法のそれは従来法に対して、60ユニットのポリグリシン分子で50%、70ユニットで30%、90ユニットで12%であり、
大幅に演算時間を短縮することができました。しかも、従来法により全系をまともに解く方法との計算誤差は殆どゼロです(<μHartree)。
|
 |
| さらに、この計算手法を用いると、単に高分子の電子状態を計算するだけではなく、
高分子の重合反応をシミュレーションしながら効率よく電子状態を計算できるので、
例えば磁性・導電性・光学特性などをもつ機能性高分子材料や生体材料を原子・分子レベルで分子設計できるようになるための強力な手段になりえます。
将来の高分子材料開発において、合成するための指針を予めコンピュータ上で候補を絞ることができ、多くの試行実験を行わずにすむので、
大幅な開発経費の削減につながるものと期待できます。また、本手法によって、
従来の量子化学計算では取り扱うことのできなかった巨大系の電子状態が短時間で計算可能となれば、
様々な条件下における蛋白質構造と機能に関する分子レベルでの解明が可能になります。種々の生体高分子の機能や役割、
生理活性現象のメカニズムを経験則ではなく、電子論に基づく一般論として明らかにすることができれば、生体機能の学問的発展に大いに貢献できます。 |